ООО "ПРОПИОНИКС"
| пн-пт с 09:00 до 18:00 | |
Рибосомальная 16S РНК, полимеразная цепная реакция (ПЦР) и секвенирование биополимеров
ОБЩИЕ СВЕДЕНИЯ О СЕКВЕНИРОВАНИИ БИОПОЛИМЕРОВ
16S рРНК, полимеразная цепная реакция (ПЦР) и секвенирование биополимеров (ДНК и РНК)
Содержание:
- 16S рPHK — удостоверение личности бактерии
- Секвенирование биополимеров
- Секвенирование РНК
- Секвенирование ДНК
- 1. Выделение нуклеиновых кислот
- 2. Разрезание и сшивание ДНК
- 3. Разделение ДНК и белков
- Электрофорез в геле
- Электроффорез в полиакриламидном геле
- Электрофорез на агарозном геле
- Саузерн блоттинг
- Денатурирующий градиентный гель-электрофорез (DGGE)
- 4. Клонирование ДНК
- BAC-клонирование
- Стратегии ПЦР-клонирования
- TA-клонирование и клонирование по тупым концам
- Основы библиотечного строительства ДНК
- Клонирование и секвенирование дробовика
- 5. Полимеразная цепная реакция (ПЦР)
- ПЦР и количественная ПЦР в реальном времени
- 6. Секвенирование ДНК по Сэнгеру
- Метод «плюс-минус»
- Метод «терминаторов»
- 7. Секвенирование нового поколения NGS
- Платформа illumina
- Технологии SLR на платформах illumina и 10XGenomic
- Пиросеквенирование
- Платформа 454 Roshe
- Пречень платформ NGS
- Технология Ion Torrent
- Нанопоровое секвенирование
- Материал в помощь (литература + курс видеолекций)
Примечание редактора: данный раздел невозможно воспринять без базовых заний о ДНК, поэтому перед чтением материала рекомендуем ознакомиться с общей информацией о ДНК - как об основе молекулярной биологии (или освежить память, если ранее тема изучалась). Материал о ДНК доступен по кнопке-ссылке:
Важнейшее правило, которое необходимо было четко уяснить после изучения структуры и свойств ДНК, и которое является одной из основ молекулярной биологии, является правило (или принцип) комплементарности:
 Принцип комплементарности - это строгое соответствие азотистых оснований в ДНК, в котором аденин соединяется с тимином двумя водородными связями (А=Т), а гуанин соединяется с цитозином тремя водородными связями (Г=Ц) (см. на рис. слева).
Принцип комплементарности - это строгое соответствие азотистых оснований в ДНК, в котором аденин соединяется с тимином двумя водородными связями (А=Т), а гуанин соединяется с цитозином тремя водородными связями (Г=Ц) (см. на рис. слева).
Такое взаимодействие играет ключевую роль в ряде фундаментальных процессов хранения и передачи генетической информации: репликации ДНК, обеспечивающей передачу генетической информации при делении клетки, транскрипции ДНК в РНК при биосинтезе белков, хранении генетической информации в двухцепочечной ДНК и процессах репарации ДНК при ее повреждении. Таким образом, принцип комплементарности лежит в основе различных взаимодействий, удвоения либо репликации молекул ДНК, по нему образуется дочерняя цепочка. При последующем делении материнской клетки каждая дочерняя получает по 1 копии молекулы ДНК. Многие методы секвенирования ДНК основаны на взаимной комплементарности цепей этой молекулы. Этот принцип лежит в основе амплификации (см. ПЦР) целевых фрагментов ДНК и их последующего секвенирования.
Итак, нуклеотидный состав ДНК подчиняется правилам Э. Чаргаффа:
1. Количество аденина (А) равно количеству тимина (Т), а количество гуанина (Г) - количеству цитозина (Ц):
А = Т, Г = Ц
2. Количество пуринов равно количеству пиримидинов:
А + Г = Т + Ц
Нуклеотидный состав РНК подобным правилам не подчиняется.
Итак, комплементарность (дополнительность) применительно к ДНК выражается в том, что напротив, каждого азотистого основания одной цепи находится строго определенное азотистое основание другой цепи, причем одно из них пурин, другое - пиримидин (сравни с рис. выше).

Новый взгляд на микромир
Развитие методов молекулярной биологии вывело ученых на новый уровень понимания процессов симбиоза человека и его микрофлоры, которые казались хорошо изученными и от дальнейшего изучения которых не ждали особых сюрпризов. Стремительный рост скорости и падение стоимости методов секвенирования ДНК (определения ее нуклеотидной последовательности) и параллельный рост мощности персональных компьютеров и развитие интернета дали возможность анализировать информацию о крупных участках геномов. После того как были расшифрованы хромосомы сотен видов отдельных бактерий, в генетике микроорганизмов появился новый подход — популяционный: анализ генов сразу всех бактерий, населяющих определенный ареал. Разумеется, население «человеческого биореактора» оказалось одной из наиболее важных для изучения микробных популяций.
Первая работа, заставившая совершенно по-новому взглянуть на кишечную микробиоту, была опубликована в 1999 году группой ученых из Национального института агрономических исследований (Франция) и Университета Ридинга (Великобритания). Авторы решили применить для исследования микробной популяции кишечника метод секвенирования генов 16S РНК.
16S рPHK — удостоверение личности бактерии

Идентификация любого штамма начинается с описания физиологических характеристик микроорганизмов. Однако, не всегда знание данных признаков позволяет с уверенностью определить систематическую принадлежность к тому или иному роду и виду.
Широко, например, известен полиморфизм бифидобактерий, что затрудняет их определение (систематику). Вилочковидная форма бифидобактерий как правило возникает на бедных средах. При выделении новых культур используют богатые среды, поэтому часто наблюдаются прямые палочки, коккобациллы, порой они образуют даже цепочки клеток. Довольно часто при первичной идентификации лактобациллы можно отнести к бифидобактериям. Описанные в литературе родоспецифичные праймеры обладают недостаточной специфичностью, что может приводить к ложноположительным результатам.
На гифке выше - Модель малой субъединицы рибосомы Thermus thermophilus. РНК показана оранжевым, белок — фиолетовым.
Чтобы избежать ложноположительных результатов при первичной идентификации штаммов, для окончательной идентификации выделенных бифидобактерий (и др. прокариот) необходим анализ секвенирования гена рибосомной 16S РНК.
На рисунке сверху: Структура гена 16S. Девять переменных областей (V1-V9) изображены зеленым цветом. Фиолетовые полосы указывают на участки гена, которые в основном используются для бактериальной классификации при амплификации и секвенировании на основе ПЦР.
Секвенирование биополимеров (белков и нуклеиновых кислот — ДНК и РНК) — определение их аминокислотной или нуклеотидной последовательности. В результате получают формальное описание первичной структуры линейной макромолекулы в виде последовательности мономеров в текстовом виде →.
16S рРНК - один из трёх основных типов рРНК, образующих основу рибосом прокариот, находятся в их малой (30S) субъединице. Константа седиментации равна 16S (единиц Сведберга); константы двух других молекул равны 5 и 23S. Длина 16S рРНК - около 1600 нуклеотидов. Из трёх типов рРНК удобнее всего анализировать 16S и 18S рРНК. У эукариот существуют аналогичные рибонуклеиновые кислоты 18S рРНК, состоящие приблизительно из 2500 нуклеотидов.
Итак, первый этап определения микроорганизмов - их культивирование на питательных средах. Но ряд микробов не желают расти ни на одной из сред (некультивируемые):
Современные методики
Изучать ранее недоступные некультивируемые бактерии и начать наводить порядок в донельзя запутанной систематике уже известных прокариот стало возможным с развитием биоинформатики и появлением современных методов молекулярной биологии - ПЦР (полимеразной цепной реакции), позволяющей из одного участка ДНК получить миллиарды точных копий, клонирования выделенных генов в бактериальных плазмидах и методик секвенирования последовательностей нуклеотидов, полученных в достаточном для анализа количестве. Идеальным маркером для идентификации микроорганизмов оказался ген, кодирующий 16S рибосомальную РНК (каждая из двух субъединиц рибосом — клеточных мастерских по синтезу белка — состоит из переплетенных молекул белков и цепочек рибонуклеиновых кислот).
Идеальный маркер
Этот ген есть в геноме всех известных бактерий и архей, но отсутствует у эукариот и вирусов, и если вы нашли характерную для него последовательность нуклеотидов - вы точно имеете дело с генами прокариот. Этот ген имеет как консервативные участки, одинаковые у всех прокариот, так и видоспецифичные. Консервативные участки служат для первого этапа полимеразной цепной реакции — присоединения исследуемой ДНК к праймерам (затравочным участкам ДНК, к которым изучаемая цепочка нуклеотидов должна присоединиться для начала анализа остальной последовательности), а видоспецифичные - для определения видов. Степень схожести видоспецифичных участков отражает эволюционное родство разных видов. Для клонирования и последующего анализа можно использовать саму рибосомальную РНК, которая в любой клетке присутствует в большем количестве, чем соответствующий ей ген. Нуклеотидные последовательности 16S рРНК всех известных бактерий и архей общедоступны. Выявленные последовательности сравнивают с имеющимися в базах данных и идентифицируют вид бактерии или объявляют ее принадлежащей к некультивируемому виду.
Новая систематика
В последнее время идет интенсивный пересмотр старой, фенотипической классификации бактерий, основанной на плохо формализуемых критериях — от внешнего вида колоний до пищевых предпочтений и способности окрашиваться разными красителями. Новая систематика опирается на молекулярные критерии (16S РНК) и только отчасти повторяет фенотипическую.
Что у нас внутри
 Кодирующие последовательности 16S РНК с помощью полимеразной цепной реакции (ПЦР) извлекали непосредственно из «окружающей среды» - 125 мг человеческого, извините, стула встраивали в плазмиды кишечной палочки (не потому, что она кишечная, а потому, что Escherichia coli - одна из любимых рабочих лошадок молекулярных биологов) и снова выделяли из культуры размножившихся бактерий. Таким образом была создана библиотека генов рибосомной 16S РНК всех микроорганизмов, находившихся в образце. После этого случайным образом было отобрано и секвенировано 284 клона. Оказалось, что только 24% полученных последовательностей 16S РНК принадлежали известным ранее микроорганизмам. Три четверти микрофлоры, находящейся в кишечнике каждого человека, больше сотни лет избегали внимания исследователей, вооруженных методами классической микробиологии! Ученые просто не могли подобрать условия для культивирования этих бактерий, потому что самые капризные обитатели кишечника отказывались расти на традиционных микробиологических средах.
Кодирующие последовательности 16S РНК с помощью полимеразной цепной реакции (ПЦР) извлекали непосредственно из «окружающей среды» - 125 мг человеческого, извините, стула встраивали в плазмиды кишечной палочки (не потому, что она кишечная, а потому, что Escherichia coli - одна из любимых рабочих лошадок молекулярных биологов) и снова выделяли из культуры размножившихся бактерий. Таким образом была создана библиотека генов рибосомной 16S РНК всех микроорганизмов, находившихся в образце. После этого случайным образом было отобрано и секвенировано 284 клона. Оказалось, что только 24% полученных последовательностей 16S РНК принадлежали известным ранее микроорганизмам. Три четверти микрофлоры, находящейся в кишечнике каждого человека, больше сотни лет избегали внимания исследователей, вооруженных методами классической микробиологии! Ученые просто не могли подобрать условия для культивирования этих бактерий, потому что самые капризные обитатели кишечника отказывались расти на традиционных микробиологических средах.
На сегодняшний день при помощи молекулярных методов установлено, что в микробиоте взрослого человека представлены 10 из 70 крупных бактериальных таксонов (дополнительно о секвенировании 16S рРНК см. здесь →).
СЕКВЕНИРОВАНИЕ БИОПОЛИМЕРОВ
Секвенирование биополимеров (белков и нуклеиновых кислот - ДНК и РНК) - определение их аминокислотной или нуклеотидной последовательности (от лат. sequentum - последовательность). В результате секвенирования получают формальное описание первичной структуры линейной макромолекулы в виде последовательности мономеров в текстовом виде. В результате секвенирования перекрывающихся участков ДНК получают последовательности участков генов, целых генов, тотальной мРНК и даже полных геномов организмов.
Рассмотрим сначала ДНК. Молекулы полимеров характеризуются первичной структурой, под которой понимается просто состав молекулы (в случае ДНК – это последовательность букв A, C, G и T, которые и составляют геном), вторичной структурой, т.е. тем, какие именно химические связи устанавливаются между этими компонентами и какие в результате получаются базовые пространственные структуры (в данном случае – двойная спираль), и третичной структурой, т.е. тем, как вторичная структура «уложена» в пространстве. Вторичная структура ДНК представляет собой двойную спираль, состоящую из четырёх разных нуклеотидов. Нуклеотиды обозначаются по содержащимся в них азотистым основаниям: аденину (A), цитозину (C), гуанину (G) и тимину (T) (есть ещё урацил, который в РНК заменяет тимин), и в дальнейшем мы всегда будем пользоваться этими буквами. В двойной спирали эти аминокислоты связаны друг с другом водородными связями, и связь устанавливается по принципу комплементарности: если в одной нити ДНК стоит A, то в комплементарной нити будет T; а если в одной нити C, то в другой будет G. Именно это позволяет относительно просто проводить репликацию (копирование) ДНК, например, при делении клетки: для этого достаточно просто разорвать водородные связи, разделив двойную спираль на нити, после чего парная нить для каждого «потомка» автоматически соберётся правильно. Важно понять, что ДНК – это две копии одного и того же «текста» из четырёх «букв»; «буквы» в копиях не идентичны, но однозначно соответствуют друг другу.
Например:
ATGCAGAACAGACGATCAGCGACACTTTA
TACGTCTTGTCTGCTAGTCGCTGTGAAAT
Было бы, конечно, удобно, если бы нам удалось аккуратно «вытянуть» одну нить ДНК и спокойно, нуклеотид за нуклеотидом, «прочесть» эту нить от начала до конца. При таком идеальном методе секвенирования (чтения ДНК) никаких хитрых алгоритмов не понадобилось бы. К сожалению, на данном этапе такое невозможно, и приходится довольствоваться результатами того секвенирования, которое есть.
Секвенирование РНК
Секвенирование РНК: Большинство экспериментов по секвенированию РНК проводятся на оборудовании, которое предназначено для секвенирования молекул ДНК. В связи с этим необходимым шагом для секвенирования РНК является создание библиотеки кДНК (комплементарной ДНК), полученной из исследуемой тотальной РНК. Каждая кДНК из такой библиотеки представляет собой фрагмент ДНК разного размера, фланкированный по обоим краям специальными адаптерами. Наличие адаптеров необходимо для последующей амплификации образцов и секвенирования. Методы создания библиотек кДНК варьируются в зависимости от конечной цели исследования и типа изучаемой РНК (РНК может различаться в размере, последовательности, структурных особенностях а также в концентрации). Перед созданием бибилиотеки кДНК, подходящей для конкретного эксперимента, необходимо ответить на следующие вопросы: 1) какие именно молекулы РНК представляют интерес; 2) как получить кДНК желаемого размера; 3) каким способом лучше присоединенить адаптерные последовательности к краям кДНК для амплификации и секвенирования.
Перед секвенированием кДНК ее необходимо амплифицировать с помощью ПЦР. Непосредственно перед проведением ПЦР можно ввести молекулярные маркеры. Эта процедура особенно актуальна, если РНК в образце изначально немного, как, например, в случае секвенирования РНК одной клетки.
Метод секвенирования РНК становится основным методом определения того, какие гены и на каком уровне экспрессируются в клетке. С помощью РНК секвенирования можно определять различия в экспрессии генов на различных стадиях развития организма или в разных тканях.
Секвенирование ДНК
Секвенирование ДНК – это общее название методов, которые позволяют установить последовательность нуклеотидов в молекуле ДНК. В настоящее время нет ни одного метода секвенирования, который бы работал для молекулы ДНК целиком; все они устроены так: сначала готовится большое число небольших участков ДНК (клонируется молекула ДНК многократно и «разрезается» её в случайных местах), а потом читается каждый участок по отдельности.
Клонирование происходит либо просто выращиванием клеток в чашке Петри, либо (в случаях, когда это было бы слишком медленно или по каким-то причинам не получилось бы) при помощи так называемой полимеразной цепной реакции. В кратком и неточном изложении работает она примерно так: сначала ДНК денатурируют, т.е. разрушают водородные связи, получая отдельные нити. Затем к ДНК присоединяют так называемые праймеры; это короткие участки ДНК, к которым может присоединиться ДНК-полимераза – соединение, которое, собственно, и занимается копированием (репликацией) нити ДНК.

Рис. 5. Схематическое изображение процесса репликации ДНК: (1) Отстающая цепь (запаздывающая нить), (2) Ведущая цепь (лидирующая нить), (3) ДНК-полимераза α (Polα), (4) ДНК-лигаза, (5) РНК-праймер, (6) Праймаза, (7) Фрагмент Оказаки, (8) ДНК-полимераза δ (Polδ), (9) Хеликаза, (10) Однонитевые ДНК-связывающие белки, (11) Топоизомераза.
На следующем этапе полимераза копирует ДНК, после чего процесс можно повторять: после новой денатурации отдельных нитей будет уже вдвое больше, на третьем цикле – вчетверо, и так далее.
Все эти эффекты достигаются в основном с помощью изменений температуры смеси из ДНК, праймеров и полимеразы; для наших целей важно, что это достаточно точный процесс, и ошибки в нём редки, а на выходе получается большое число копий участков одной и той же ДНК. Разные методы секвенирования отличаются друг от друга не методами клонирования, а тем, как потом прочесть получившийся «суп» из многочисленных копий одной и той же ДНК...

Примечание редактора
Если имеется желание ознакомиться с темой секвенирования более детально, а не в обзорном порядке, то в данном разделе предусмотрен т.н. "материал в помощь", где в трех литературных источниках и в видеолекциях можно найти всю интересующую информацию к темам этой страницы.
1. Выделение ДНК и РНК
Выделение нуклеиновых кислот
Практически все научные исследования в области молекулярной биологии на той или иной стадии включают этап выделения нуклеиновых кислот. Выделенные нуклеиновые кислоты затем используют в ПЦР, секвенировании и для множества других задач, причем технологии выделения различаются не только по принципу своего действия, но и в зависимости от типа биоматериала и последующего применения экстракта.
Впервые нуклеиновые кислоты пытались выделить в середине XIX века, когда ещё практически ничего не было известно об этих молекулах. Однако с момента открытия структуры и свойств ДНК технологии её выделения непрерывно модифицируются и совершенствуются. В данной статье рассматриваются самые распространенные, а также прогрессивные методики, используемые для экстракции нуклеиновых кислот.
Итак, выделение ДНК и РНК - важный шаг подготовки проб для выполнения различных задач в микробиологии, биотехнологии, биохимии, медицинской диагностике и т.д.. Амплификация, проведение обратной транскрипции, детектирование накопления продуктов амплификации методом ПЦР в реальном времени, клонирование, секвенс, гибридизация, синтез ДНК и т.д., не могут быть выполнены непосредственно на биологических образцах без предварительного выделения (экстракции) нуклеиновых кислот (НК) и их последующей очистки.
На сегодняшний день имеется множество специализированных методик, которые могут использоваться для выделения нуклеиновых кислот с высокой степенью очистки. Наиболее известные из них относятся к методикам осаждения НК на суспензионный носитель и выделения НК на колонках. Однако набирают популярность и другие методы, о которых будет сказано позднее.
1. В зависимости от того, из какого организма выделяют НК используют различные методы разрушения клеток:
- Для разрушения клеток бактерий используют химические вещества, разрушающие клеточную стенку бактерий – ЭДТА, лизоцим, ультразвук, гомогенизация и др. Для лизиса клеток и денатурации белков часто используется детергент додецилсульфат натрия или гуанидинизотиоцианат.
- Разрушение клеток животных и человека не вызывает сложностей: используют гомогенизацию, обработку SDS (додецилсульфатом натрия), либо клетки обрабатывают протеиназами.
- Для разрушения клеточных стенок растений – ферменты, разрушающие целлюлозу, замораживание в жидком азоте и последующее механическое разрушение клеток и др.
Часто используют обработку детергентами, растворяющих мембраны клеток, и хелатирующими агентами, подавляющих действие клеточных нуклеаз за счѐт связывания двухвалентных катионов.
2. Отделение нуклеиновых кислот от белков
- Депротеинизацию клеточного лизата часто осуществляют с помощью фенола и хлороформа (белки переходят в фазу растворителя). Молекулярщики часто подразумевают смесь водонасыщенного фенола с хлороформом 1:1, а не кристаллическое вещество. В смеси с хлороформом фенол работает эффективнее, а изоамиловый спирт гасит пенообразование.
- Часто белки разрушают протеиназами, например, протеиназой К; центрифугированием для удаления денатурированных белков и фрагментов клеточных органелл.
- Ряд современных методов предусматривает осаждение ДНК на гранулах силикагеля, центрифугирование и последующую элюцию ДНК с гранул в раствор.
- Некоторые коммерческие наборы предусматривают сорбцию ДНК на мембранах или ионообменных сорбентах.
Когда нуклеиновые кислоты остаются в водном растворе:
ДНК осаждают из раствора этанолом и после центрифугирования растворяют осадок в буферном растворе. Вместе с ДНК частично выделяется и РНК, от которой избавляются с помощью фермента РНКазы.
Концентрацию полученной нуклеиновой кислоты, а также наличие примесей (белки) обычно определяют спектрофотометрически по поглощению на А260 нм. Максимум поглощения белка приходится на 280 нм. Одна (каждая) оптическая единица соответствует концентрации ДНК в 50 мкг/мл.
Для оценки чистоты препарата ДНК, свободного от РНК, проводят измерения оптической плотности раствора при длинах волн 260, 280 и 235 нм, т.е. на максимумах поглощения растворов ДНК, белков и полисахаридов, соответственно. Значение соотношения А260/280 для чистой ДНК должно быть больше 1,8. Значение А260/235 должно быть больше, чем 2,2.
Пять популярных методик выделения нуклеиновых кислот
Выделение фенол-хлороформом

Рис. 7. Схема протокола выделения фенол-хлороформным методом.
Первое упоминание об использовании этого метода встречается в статье 1967 года, и с тех пор эта технология является одним из самых распространённых способов выделения нуклеиновых кислот.
Суть методики заключается в смешивании клеточного лизата с фенолом, хлороформом и изоамиловым спиртом в пропорции 25:24:1 и последующем перемешивании и центрифугировании смеси. После проведения этих манипуляций получается раствор с двумя фазами: водной и органической, причем все липиды и жиры находятся в органической (нижней) фазе, белки — на границе фаз, а нуклеиновые кислоты — в водной (верхней) фазе (Рис.7). Для повышения чистоты экстракта эти действия повторяют несколько раз. Если раствор будет иметь низкий pH, то ДНК перейдёт в органическую фазу, а РНК останется в водной фазе, что позволяет выделять РНК отдельно от ДНК.
Данный метод используется повсеместно, поскольку он не требует дополнительного сложного оборудования и имеет невысокую стоимость. Однако нуклеиновые кислоты, полученные таким образом, обладают невысоким качеством и зачастую требуют дополнительной очистки. Также эта технология имеет существенно меньший выход нуклеиновых кислот в сравнении с другими методиками.
Помимо качества экстракта, этот метод обладает ещё несколькими недостатками: он требует сложных манипуляций, которые могут привести к контаминации и потере образца, а сам процесс трудно автоматизировать. Также весь протокол занимает достаточно много времени.
Выделение на спин-колонках

Рис. 8. Схема протокола выделения на спин-колонках.
Технология выделения на спин-колонках — это усовершенствованный метод экстракции на частичках силики, предложенный американскими учёными в 1979 году. Они продемонстрировали, что в щелочных условиях и при повышенных концентрациях соли ДНК связывается с силикатами, и это позволяет отделить все остальные компоненты клетки от частиц силики со связанной ДНК. Спин-колонки сконструированы таким образом, что при нанесении клеточного лизата на колонку и последующем центрифугировании ДНК остаётся на колонке, а всё лишнее проходит сквозь неё (Рис.8). Затем ДНК промывают несколько раз и элюируют в пробирку для сбора образца.
Преимущества такого метода заключаются в повышенной чистоте и хорошем качестве выделенных нуклеиновых кислот, высокой воспроизводимости и простоте по сравнению с выделением фенол-хлороформом. Однако также большое количество манипуляций может привести к контаминации, а выделение коротких фрагментов ДНК на спин-колонках может быть затруднено.
Экстракция на спин-колонках может занять от 20 минут в зависимости от биоматериала и сложности его лизиса.
На рынке этот метод представлен большим разнообразием наборов от таких производителей, как Qiagen, Analytik Jena, NEB, ThermoFisher и других. Стоимость одного выделения здесь значительно выше, чем у предыдущего метода, поскольку на каждую реакцию необходима своя колонка, несколько пробирок для сбора фильтрата и элюата и, конечно, реагенты.
Выделение на магнитных частицах

Рис. 9. Схема протокола выделения на магнитных частицах.
Спустя 20 лет после появления метода выделения на спин-колонках начинает набирать популярность более быстрый способ выделения на магнитных частицах 3. Технология этого способа выделения основана на связывании нуклеиновой кислоты с веществом, покрывающим магнитные частицы (целлюлоза, сефадекс, сефакрил, dT-олигонуклеотиды, специфичные олигонуклеотиды и др.). К клеточному лизату добавляют такие магнитные частицы и перемешивают для связывания ДНК с ними. После этого пробирку ставят в магнитный штатив или подносят к магниту, фиксируя таким образом твердую фазу. После отбора супернатанта нуклеиновые кислоты на частицах промывают и элюируют (Рис.9).
Этот метод имеет те же преимущества, что и выделение на спин-колонках, но для экстракции на магнитных частицах не требуется сложное лабораторное оборудование (например, центрифуга). Более того, процесс выделения на магнитных частицах легко автоматизировать, и многие автоматические станции выделения основаны именно на этой методике. Однако здесь также присутствует риск контаминации и потерь образца.
Данный протокол выделения займет немного меньше времени благодаря отсутствию этапов центрифугирования, но количество манипуляций будет примерно таким же. Также стоимость одной реакции обычно выше, чем при выделении на колонках, а панели наборов предоставляют Qiagen, Analytik Jena, ThermoFisher и другие.
Умное выделение (Smart Extraction)

Рис. 10. Схема протокола умного выделения. Протокол основан на принципе работы наборов для выделения компании Analytik Jena.
Методики выделения нуклеиновых кислот не перестают совершенствоваться: в 2005 году специалисты из компании Analytik Jena доказали, что для связывания нуклеиновых кислот с неорганической твёрдой фазой можно использовать не только хаотропные соли, но и смесь из хаотропных и нехаотропных солей с низкой ионной силой, эту технологию они назвали Dual Chemistry.
Позднее они усовершенствовали технологию Dual Chemistry при помощи немагнитных частиц с «умной» поверхностью. Для выделения используются специальные наконечники с этими частицами, которые надеваются на дозатор. При заборе клеточного лизата в наконечник нуклеиновая кислота из раствора связывается с частицами, затем следуют этапы промывки и элюции, в результате чего получается очищенная нуклеиновая кислота высокого качества (Рис.10).
Эта технология значительно ускоряет процесс выделения, а благодаря особенностям «умной» поверхности выход и качество нуклеиновых кислот значительно превосходит все предыдущие методы. Данный способ экстракции очень легко автоматизировать, ведь носики со связывающими частицами подходят как для обычных лабораторных дозаторов, так и для различных автоматических станций выделения нуклеиновых кислот.
Ферментативное температурно-зависимое выделение

Рис. 11. Схема протокола ферментативного температурно-зависимого выделения. Протокол основан на принципе работы наборов для выделения компании MicroGEM. * — несмотря на невысокую степень очистки, образец отлично подходит для дальнейшего использования в ПЦР, секвенировании и STR.
Все вышеперечисленные методики имеют общую лимитирующую стадию — этап лизиса. Во всех технологиях используется SDS и протеиназа K для разрушения клеточных стенок и высвобождения нуклеиновых кислот. SDS является ингибитором ПЦР, именно поэтому необходимы множественные стадии промывки, которые повышают риск контаминации и приводят к потерям образца. Также более сложные для лизиса образцы могут требовать дополнительную долгую и трудозатратную пробоподготовку.
Специалисты из новозеландской компании MicroGEM ликвидировали проблемы, связанные с длительным и сложным лизисом и использованием вредных химикатов, благодаря применению очень эффективной термофильной протеиназы EA1 вместе с мезофильными гидролазами.
Процесс ферментативного температурно-зависимого выделения начинается со смешивания буфера и ферментов с образцом. При последующей инкубации при комнатной температуре гидролазы деградируют клеточные стенки. После этого пробирку нагревают до 75°C, что активирует работу протеиназы EA1, которая разрушает все белки и высвобождает нуклеиновые кислоты. Последующее нагревание до 95°C дезактивирует EA1, и после этого образец готов для дальнейшего исследования (Рис.11). Для особо загрязненных образцов вроде почвы или растений можно добавить этап очистки на колонке для избавления от ингибиторов.
Данная технология оптимальна для работы с малым количеством биоматериала, поскольку нет потерь нуклеиновых кислот. Также эту методику легко автоматизировать, она самая быстрая среди всех упомянутых способов выделения (от 7 минут) и включает меньше всего манипуляций. Стоимость одной реакции невысока, поскольку кроме реагентов не требуется никаких специальных расходных материалов.
2. Разрезание и сшивание ДНК

Рестрикция и рестриктазы
Разрезание ДНК с помощью рестрикционных эндонуклеаз
Одним из первых и важнейших из шагов молекулярной биологии стала возможность разрезать молекулы ДНК, причем в строго определенных местах. Этот метод был изобретен при изучении в 1950—1970-е годы такого феномена: некоторые виды бактерий при добавлении в среду чужеродной ДНК разрушали ее, в то время, как их собственная ДНК оставалась невредимой. Оказалось, что они для этого используют ферменты, позднее названные рестрикционными нуклеазами или рестриктазами. Важным свойством каждого подобного фермента является его способность разрезать строго определенную - целевую - последовательность нуклеотидов ДНК.
Рестриктазы не воздействуют на собственную ДНК клетки, поскольку нуклеотиды в целевых последовательностях модифицированы так что, рестриктаза не может с ними работать (Правда, иногда, наоборот, они могут разрезать только модифицированные последовательности - для борьбы с теми, кто модифицирует ДНК, защищаясь от вышеописанных рестриктаз). Из-за того, что целевые последовательности бывают различной длины, частота встречаемости их в молекулах ДНК варьирует чем: длиннее необходимый фрагмент, тем меньше вероятность его появления. Соответственно, образующиеся при обработке различными рестриктазами фрагменты ДНК будут иметь различную длину.
 Рисунок слева. Сайты рестрикции. Сверху — целевая последовательность рестриктазы SmaI, при работе которой образуются «тупые» концы. Снизу — целевая последовательность рестриктазы EcoRI, при работе которой образуются «липкие» концы.
Рисунок слева. Сайты рестрикции. Сверху — целевая последовательность рестриктазы SmaI, при работе которой образуются «тупые» концы. Снизу — целевая последовательность рестриктазы EcoRI, при работе которой образуются «липкие» концы.
Итак, рестриктазы – это группа ферментов, относящихся к классу гидролаз, катализирующих гидролиз фосфодиэфирных связей чужеродных ДНК в большинстве прокариотических и некоторых других организмах и выполняющие тем самым «иммунную» функцию – системы рестрикции-модификации. Для исследований их выделяют преимущественно из прокариотических клеток.
Данные ферменты, «узнающие» определенные последовательности (сайты рестрикции) в двухцепочечной ДНК, расщепляют нуклеиновые кислоты в середине молекулы. При этом каждая рестриктаза узнаѐт определѐнный участок ДНК длиной от четырѐх пар нуклеотидов и расщепляет нуклеотидную цепь внутри участка узнавания или вне него.
Различают 3 класса рестриктаз:
- Рестриктазы 1 типа - разрезают двухцепочную молекулу ДНК неподалѐку от опознанной последовательности нуклеотидов в произвольной точке и само место разреза не строго специально.
- Рестриктазы 2 типа - разрезают двойную спираль ДНК в определѐнной фиксированной точке внутри опознанной последовательности. Рестриктазы этого типа - узнают палиндромальные последовательности, которые обладают центральной осью и считываются одинаково в обе стороны от оси симметрии.
- Рестриктазы 3 (промежуточного) типа узнают нужную последовательность и разрезают двухцепочную молекулу ДНК, отступив определѐнное число нуклеотидных пар от еѐ конца (или в нескольких точках на разном удалении от сайта узнавания). При этом образуются фрагменты ДНК либо с ровными (тупыми) концами, либо с выступающими (липкими) 5'- или 3'-концами. Эти рестриктазы узнают асимметричные сайты.
Также рестриктазы делят на мелко- и крупнощепящие. Мелкощепящие рестриктазы узнают тетрануклеотид (последовательность из 4-х пар оснований) и вносят в молекулы гораздо больше разрывов, чем крупнощепящие, узнающие последовательность из шести нуклеотидных пар.
Рестрикционный анализ ДНК
Для каждого фермента рестрикции существуют оптимальные условия реакции, которые приводятся в описании, прилагаемом фирмой-изготовителем. Основные переменные параметры – это температура инкубации и состав буфера. К температурному режиму предъявляются достаточно жесткие требования, тогда как различия между буферами чаще всего лишь незначительны.
Рестрикционный анализ ДНК широко используется в молекулярно-биологических исследованиях и прикладных работах и является одним из наиболее важных инструментов при изучении ДНК. При помощи эндонуклеаз рестрикции можно исследовать ДНК различных вирусов, бактерий, животных, растений.
Как правило, продукты расщепления ДНК анализируются с помощью гель-электрофореза в агарозном или акриламидном геле, а полученная таким образом картина разделения фрагментов ДНК в виде определенного, отличающегося для разных ферментов, набора полос и является результатом рестрикционного анализа той или иной ДНК.
Короткие фрагменты мигрируют намного быстрее, чем длинные. При сравнительно высокой концентрации агарозы большие фрагменты вообще не могут проникнуть в гель. В процессе миграции рестрикционные фрагменты не деградируют, их можно вымывать в виде биологически активных двухцепочечных молекул. При окрашивании гелей красителями, связывающимися с ДНК, выявляется набор полос, каждая из которых отвечает рестрикционному фрагменту, молекулярную массу которого можно определить, проведя калибровку с помощью ДНК с известными молекулярными массами (подробнее см. об электрофорезе).
При использовании нескольких эндонуклеаз рестрикции на одном образце можно составлять рестрикционные карты. Располагая такой информацией, можно идентифицировать на ДНК биологически важные участки. Поскольку рестрикционная карта отражает расположение определенной последовательности нуклеотидов в данном участке, сравнение таких карт для двух или более родственных генов позволяет оценить гомологию между ними. Анализируя рестрикционные карты, можно сравнивать определенные участки ДНК разных видов животных без определения их нуклеотидной последовательности. Таким образом, например, было установлено, что хромосомные участки, кодирующие цепи гемоглобина у человека, орангутанга и шимпанзе сохранились в практически неизменном виде в течение последних 5 - 10 млн. лет.
Метод рестрикционного картирования позволяет увидеть крупные генетические изменения, такие как делеции или инсерции. При этом происходит уменьшение или увеличение рестрикционных фрагментов, а также исчезновение или возникновение сайтов рестрикции.
Лигирование
Сшивание молекул ДНК посредством ферментов ДНК-лигаз

Рисунок 14. Реакция Т4 ДНК-лигазы
Для создания новых молекул ДНК, разумеется, кроме разрезания, необходима еще и возможность сшивания двух цепей. Это делают с помощью ферментов, называемых ДНК-лигазами, которые сшивают сахаро-фосфатный остов двух цепей ДНК. Поскольку по химическому строению ДНК не отличается у разных организмов, можно сшивать ДНК из любых источников, и клетка не сможет отличить полученную молекулу от своей собственной ДНК.
Наиболее распространенным ферментом, используемым для лигирования, является T4 ДНК-лигаза, которая связывает (катализирует образование фосфодиэфирной связи) концы ДНК между 5'-фосфатными и 3'-OH (гидроксильными) группами. Рекомбинантный фермент выделен из штамма кишечной палочки E. coli, несущего клонированный ген лигазы бактериофага T4. Фермент использует АТФ в присутствии Mg2+ в качестве кофактора. Для проведения реакции T4 ДНК-лигазы АТФ, Mg2+ и ДТТ обычно подаются в реакционный буфер (Рис. 14). Для улучшения результатов лигирования, общая рекомендация заключается в создании нескольких реакций с различными вставками: вектор молярных соотношений, как правило, в диапазоне от 1:1 до 5:1. Для менее эффективных лигирований, как и для фрагментов ДНК с тупыми концами, часто рекомендуется добавление инертных макромолекул, таких как полиэтиленгликоль (ПЭГ), чтобы увеличить эффективную концентрацию компонентов реакции и тем самым повысить эффективность лигирования.
3. Разделение молекул ДНК и белков
Метод гель-электрофореза
Электрофорез - это движение дисперсных частиц относительно жидкости под действием пространственно однородного электрического поля.
Часто приходится иметь дело со смесью молекул ДНК разной длины. Например, при обработке химически выделенной из организма ДНК рестриктазами как раз получится смесь фрагментов ДНК, причем их длины будут различаться.
Поскольку любая молекула ДНК в водном растворе отрицательно заряжена, появляется возможность разделить смесь фрагментов ДНК различных размеров по их длине с помощью электрофореза. ДНК помещают в гель (обычно, агарозный для относительно длинных и сильно отличающихся молекул или полиакриламидный для электрофореза с высоким разрешением), который помещают в постоянное электрическое поле. Из-за этого молекулы ДНК будут двигаться к положительному электроду (аноду), причем их скорости будут зависеть от длины молекулы: чем она длиннее, тем сильнее ей мешает двигаться гель и, соответственно, тем ниже скорость. После электрофореза смеси фрагментов разных длин в геле образуют полосы, соответствующие фрагментам одной и той же длины. С помощью маркеров (смесей фрагментов ДНК известных длин) можно установить длину молекул в образце
Физический принцип метода заключается в следующем. Находящиеся в буферном растворе макромолекулы обладают некоторым суммарным электрическим зарядом, величина и знак которого зависят от рН среды. Если через этот раствор, заключенный в канал из изолирующего материала начать пропускать электрический ток, то вдоль канала установится определенный градиент напряжения, т.е. сформируется электрическое поле. Его напряженность измеряется разностью потенциалов по концам рабочего канала (или его участка), отнесенной к его длине (В/см). Под действием поля макромолекулы в соответствии со своим суммарным зарядом мигрируют в направлении катода или анода, причем их трение об окружающую среду ограничивает скорость миграции. В зависимости от величины заряда и размеров молекулы приобретают разные скорости, и в этом – сущность процесса электрофореза. Постепенно исходный препарат, состоявший из различных молекул, разделяется на зоны одинаковых молекул, мигрирующих с одной и той же скоростью. Со временем эти зоны распределяются по длине канала.
В современных приборах рабочий канал заполняют гелем. Достаточно чистая и хорошо смачиваемая (гидрофильная) пространственная сетка геля удерживает жидкость от вытекания и препятствует конвекции. Вместе с тем, используемые гели содержат очень много жидкости (80-99,5%), в которой (т.е. в рабочем буфере) и мигрируют макромолекулы. Наличие сетки геля вносит важную дополнительную деталь в картину электрофоретической миграции. Теперь фракционируемые макромолекулы любых размеров неизбежно сталкиваются с нитями полимера, образующего сетку геля, что увеличивает эффективное трение о среду, а следовательно, снижает скорость движения молекул. Очевидно, что препятствия для миграции становятся особенно серьезными, если средний диаметр пространственных ячеек геля оказывается соизмерим с размерами макромолекул. В этом случае решающее влияние на электрофоретическую подвижность различных макромолекул и степень разделения оказывает соотношение их линейных размеров. Возможна даже такая ситуация, когда особенно крупные молекулы нуклеиновых кислот вообще не смогут «протиснуться» через поры геля и их миграция прекратится.
В настоящее время почти исключительно используются полиакриламидные гели (ПААГ) и гели агарозы. Варьируя концентрацию полимера, можно получать гели с очень широким диапазоном размеров пор. Кроме того, можно изменять электрические заряды макромолекул путем вариации рН буфера, а их конфигурацию путем введения в буфер денатурирующих агентов или детергентов. Все это придает методу электрофореза исключительную гибкость. Но есть, разумеется, и свои проблемы. Разделяемые макромолекулы все же находятся в растворе, поэтому возможна их диффузия, приводящая к размыванию зон. Это тем более серьезно, что протекание через жидкость электрического тока неизбежно связано с выделением тепла. К счастью, крупные молекулы нуклеиновых кислот диффундируют не слишком быстро.
Для визуализации результатов электрофореза проводят окрашивание зон путем вымачивания геля в растворе красителя, прочно связывающегося с нуклеиновой кислотой. Излишек красителя удаляют, а гель облучают ультрафиолетом, под действием которого связавшийся с двунитевой ДНК краситель флуоресцирует.
А) Электрофорез в полиакриламидном геле

Электрофорез в полиакриламидном геле (ПААГ или PAGE) - метод, широко используемый для разделения биологических макромолекул в соответствии с их электрофоретической подвижностью. Подвижность является функцией длины, конформации и заряда молекулы.
Как и во всех формах гель-электрофореза, молекулы могут работать в своем естественном состоянии, сохраняя структуру молекул более высокого порядка, или может быть добавлен химический денатурант, чтобы удалить эту структуру и превратить молекулу в неструктурированную линейную цепь, подвижность которой зависит только от ее длины и отношение массы к заряду.
Таким образом, разделяют т.н. нативный ПААГ-электрофорез (при котором разделяемые биологические макромолекулы в процессе электрофореза остаются в нативном состоянии) и денатурирующий ПААГ-электрофорез (при котором пробы предварительно денатурируют, в случае нуклеиновых кислот используют непродолжительное нагревание пробы с формамидом либо глиоксалем, для денатурации белков обычно используют кипячение пробы в буфере, содержащем сильный ионный детергент (обычно додецилсульфат натрия SDS).
Базовые приготовления
Образцы могут представлять собой любой материал, содержащий белки. Они могут быть получены биологически, например, из прокариотических или эукариотических клеток, тканей, вирусов, проб окружающей среды или очищенных белков. Образец для анализа необязательно смешивают с химическим денатурантом, обычно SDS для белков. SDS - это анионный детергент, который денатурирует вторичные и недисульфидно-связанные третичные структуры и дополнительно придает отрицательный заряд каждому белку пропорционально его массе. Нагревание образцов по крайней мере до 60 ° C дополнительно способствует денатурации.
Приготовление акриламидных гелей
Гели обычно состоят из акриламида, бисакриламида, необязательного денатурирующего вещества (SDS) и буфера с отрегулированным pH. Раствор можно дегазировать под вакуумом, чтобы предотвратить образование пузырьков воздуха во время полимеризации. Источник свободных радикалов и стабилизатор, такой как персульфат аммония и TEMED, добавляются для инициирования полимеризации.
Реакция полимеризации создает гель из-за добавленного бисакриламида, который может образовывать поперечные связи между двумя молекулами полиакриламида. Гели, как правило, полимеризуются между двумя стеклянными пластинами в гелеобразователе, с гребнем, вставленным вверху для создания лунок для образца. После того, как гель полимеризован, «расческа» может быть удалена, и гель готов для электрофореза.
Электрофорез
В PAGE используются различные буферные системы в зависимости от природы образца и цели эксперимента. Буферы, используемые на аноде и катоде, могут быть одинаковыми или разными. Электрическое поле воздействует на гель, заставляя отрицательно заряженные белки мигрировать через гель от отрицательного электрода (катода) к положительному электроду (аноду). В зависимости от их размера каждая биомолекула движется по-разному через матрицу геля: маленькие молекулы легче проникают через поры в геле, в то время как более крупные имеют большую сложность. Гель обычно работает в течение нескольких часов, хотя это зависит от напряжения, приложенного к гелю; Миграция происходит быстрее при более высоких напряжениях, но эти результаты обычно менее точны, чем при более низких напряжениях. По истечении заданного времени биомолекулы мигрируют на разные расстояния в зависимости от их размера. Меньшие биомолекулы движутся дальше вниз по гелю, в то время как более крупные остаются ближе к точке происхождения. Следовательно, биомолекулы могут быть разделены примерно в соответствии с размером, который зависит в основном от молекулярной массы в денатурирующих условиях, но также зависит от конформации высшего порядка в нативных условиях.
Результаты: ПААГ чаще всего окрашивают с помощью кумасси бриллиантового синего R-250 или серебряного красителя), что позволяет визуализировать отделенные белки, или подвергают дальнейшей обработке (например, Вестерн-блоттингом). После окрашивания биомолекулы разных видов появляются в виде отдельных полос внутри геля. Для калибровки геля и определения приблизительной молекулярной массы неизвестных биомолекул путем сравнения пройденного расстояния относительно маркера обычно используют маркеры размера молекулярной массы с известной молекулярной массой на отдельной дорожке в геле.
Кроме «обычного» электрофореза в пластине из геля, в некоторых случаях используют капиллярный электрофорез, который проводят в очень тонкой трубочке, наполненной гелем (обычно полиакриламидным). Разрешающая способность такого электрофореза значительно выше: с его помощью можно разделять молекулы ДНК, отличающиеся по длине всего на один нуклеотид. Об одном из важных приложений такого метода читайте в описании метода секвенирования ДНК по Сэнгеру.
Б) Электрофорез ДНК в агарозном геле

Рис. 17. Элекрофорез в агарозном геле
Самым популярным методом электрофореза с гелем является использование агарозного геля. Именно этот гель, как среду с определенным рН, используют в целях разделения, очищения и идентификации отдельных фрагментов ДНК. Почему эта методика стал столь популярна в современной генетике? Не все лабораторные методы могут разделить некоторые фрагменты ДНК, что мешает получить 100 %-нтный результат исследования. Гель электрофорез помогает выделить и разделить фрагменты дезоксирибонуклеиновой кислоты. За счет трений материалов, образующих гель, формируется «молекулярное сито», что помогает дифференцировать молекулы в соответствии с размером и зарядом. Скорость движения заряженных частиц ДНК через образованные поры в электрическом поле зависят от нескольких факторов:
- Силы образованного электрического поля;
- Диаметра и форм молекул;
- Относительной степени «боязни» воды образцов;
- Температурной кривой буфера и ионной силы.

Рисунок 18. Электрофорез в агарозном геле с использованием бромистого этидия для визуализации результатов в ультрафиолете (слева). Вторая слева дорожка-маркер с известными длинами фрагментов. Справа - Установка для проведения электрофореза в геле.
Визуализовать результаты фореза можно двумя способами. Первый, наиболее часто используемый в последнее время - добавление в гель веществ флуоресцирующих, в присутствии ДНК (традиционно использовался довольно токсичный бромистый этидий; в последнее время в обиход входят более безопасные вещества). Бромистый этидий светится оранжевым светом при облучении ультрафиолетом, причем при связывании с ДНК интенсивность свечения возрастает на несколько порядков. Другой метод заключается в использовании радиоактивных изотопов, которые необходимо предварительно включить в состав анализируемой ДНК. В этом случае на гель сверху кладут фотопластинку, которая засвечивается над полосами ДНК за счет радиоактивного излучения (этот метод визуализации называют авторадиографией)
Выявление определенной последовательности ДНК в смеси. Саузерн блоттинг

Рис. 19. Саузерн-блоттинг (от англ. Southern blot) — метод, применяемый в молекулярной биологии для выявления определённой последовательности ДНК в образце. Метод Саузерн-блоттинга сочетает электрофорез в агарозном геле для фракционирования ДНК с методами переноса разделённой по длине ДНК на мембранный фильтр для гибридизации.
С помощью электрофореза можно узнать размер молекул ДНК в растворе, однако он ничего не скажет о последовательности нуклеотидов в них. С помощью гибридизации ДНК можно понять, какая из полос содержит фрагмент со строго определенной последовательностью. Гибридизация ДНК основана на образовании водородных связей между двумя цепями ДНК, приводящем к их соединению.
Сначала необходимо синтезировать ДНК-зонд, комплементарный той последовательности, которую мы ищем. Он обычно представляет собой одноцепочечную молекулу ДНК длиной 10–1000 нуклеотидов. Из-за комплементарности зонд свяжется с необходимой последовательностью, а за счет флуоресцентной метки или радиоизотопов, встроенных в зонд, результаты можно увидеть.
Для этого используют процедуру, называемую Саузерн-блоттинг или перенос по Саузерну, названную по имени ученого, ее изобретшего (Edwin Southern). Первоначально смесь фрагментов ДНК разделяют с помощью электрофореза. На гель сверху кладут лист нитроцеллюлозы или нейлона, и разделенные фрагменты ДНК переносятся на него за счет блоттинга: гель лежит на губке в ванночке с раствором щелочи, который просачивается через гель и нитроцеллюлозу за счет капиллярного эффекта от бумажных полотенец, сложенных сверху. Во время просачивания щелочь вызывает денатурацию ДНК, и на поверхность пластины нитроцеллюлозы переносятся и закрепляются там уже одноцепочечные фрагменты. Лист нитроцеллюлозы аккуратно снимают с геля и обрабатывают радиоактивно меченной ДНК-пробой, специфичной к необходимой последовательности ДНК. Лист нитроцеллюлозы тщательно отмывают, чтобы на нем остались только те молекулы пробы, которые гибридизовались с ДНК на нитроцеллюлозе. После авторадиографии ДНК, с которой гибридизовался зонд, будет видна как полосы на фотопластинке (рис. ).

Рис. 20. Схема проведения Саузерн-блоттинга
Адаптация этой методики для определения специфических последовательностей РНК называется, в противоположность Саузерн-блоттингу, норзерн-блоттингом (northern blotting: southern по-английски означает «южный», а northern — «северный»). В этом случае проводят электрофорез в геле с молекулами мРНК, а в качестве зонда выбирают одноцепочечную молекулу ДНК или РНК.
дополнительная информация.
Денатурирующий градиентный гель-электрофорез (DGGE)
Выше мы рассмотрели основные принципы работы гель-электрофореза. Однако все чаще в литературе, посвященной исследованиям по секвенированию ДНК, можно встретить информацию об использовании метода ДГЭ или денатурирующего градиентного гель-электрфореза. В частности упоминается о т.н. ПЦР-ДГЭ. По существу, ПЦР-ДГЭ включает амплификацию ДНК с помощью родоспецифических праймеров, которые нацелены на последовательности 16S рДНК, и последующую дифференцировку этой ДНК на денатрурирующем геле.
Денатурирующий градиентный гель-электрофорез (DGGE или Denaturation Gradient Gel Electrophoresis) работает путем нанесения небольшого образца ДНК (или РНК) на электрофорезный гель, содержащий денатурирующий агент. Обнаружено, что определенные денатурирующие гели способны индуцировать расплавление ДНК на различных стадиях. В результате этого плавления ДНК распространяется по гелю и может быть проанализирована на отдельные компоненты, даже такие небольшие, как 200-700 пар оснований.
Уникальность метода DGGE заключается в том, что по мере того, как ДНК подвергается все более экстремальным условиям денатурации, расплавленные нити полностью распадаются на отдельные нити. Процесс денатурации на денатурирующем геле очень резкий (большинство фрагментов плавятся в пошаговом процессе). Дискретные части или домены фрагмента внезапно становятся одноцепочечными в очень узком диапазоне денатурирующих условий. Это позволяет различать различия в последовательностях ДНК или мутации различных генов: различия в последовательности фрагментов одинаковой длины часто приводят к тому, что они частично плавятся в разных положениях градиента и поэтому "останавливаются" в разных положениях геля.
На чем основан метод DGGE? Метод денатурирующего градиентного гель-электрофореза основан на зависимости свойств плавления (или денатурации) небольших двухнитевых молекул ДНК от их нуклеотидной последовательности, а точнее - от соотношения А-Т- и G-C-пар в исследуемых фрагментах. Объясняется это тем, что G-C-связь более прочна по сравнению со связью между нуклеотидами А и Т. Подобные различия в динамике плавления могут быть выявлены путем сравнения подвижности нормальных и мутантных двухнитевых фрагментов ДНК при их электрофорезе в денатурирующих условиях.
Градиент денатурации достигается разницей температур, различной концентрацией мочевины или формальдегида в гелях. При этих условиях одинаковые по величине двухнитевые молекулы ДНК, отличающиеся по нуклеотидной последовательности, денатурируют по-разному. Разработан компьютерный алгоритм, позволяющий предсказывать характер плавления в зависимости от нуклеотидной последовательности.
При электрофорезе амплифицированных двухнитевых фрагментов ДНК в геле с линейно возрастающим градиентом концентраций денатурирующих агентов плавление нитей ДНК происходит в строго специфичной для данной последовательности области, эквивалентной температуре плавления, т. е. такой температуре, при которой каждая пара оснований с 50 % вероятностью может соединиться или разойтись. После начала плавления продвижение двухнитевого фрагмента ДНК в геле резко замедляется вследствие сложной пространственной конфигурации молекул, причем эта задержка будет длиться до тех пор, пока не наступит полная денатурация ДНК. В результате происходит разделение фрагментов ДНК, различающихся по нуклеотидному составу.
4. Клонирование ДНК

Молекулярное клонирование - это совокупность экспериментальных методов в молекулярной биологии, которые используются для сборки рекомбинантных молекул ДНК и направления их репликации в организме хозяина. Использование слова клонирование относится к тому факту, что метод включает репликацию одной молекулы для получения популяции клеток с идентичными молекулами ДНК. Молекулярное клонирование обычно использует последовательности ДНК от двух различных организмов: вид, который является источником ДНК, подлежащей клонированию, и вид, который будет служить в качестве живого хозяина для репликации рекомбинантной ДНК.
Мы уже знаем, каким образом можно разрезать геном на части (а их сшивать с произвольными молекулами ДНК), разделять полученные фрагменты по длине и с помощью гибридизации выбрать необходимый. Теперь настало время узнать, как, скомбинировав эти методы, мы можем клонировать участок генома (например, определенный ген). В геноме любой ген занимает крайне маленькую длину (по сравнению со всей ДНК клетки). Клонирование ДНК буквально означает создание большого числа копий определенного ее фрагмента. Именно за счет такой амплификации мы получаем возможность выделить участок ДНК и получить его в достаточном для изучения количестве.
Каким образом разделить фрагменты ДНК по длине и идентифицировать нужный — было упрощенно рассказано выше. Теперь надо понять, каким образом можно копировать необходимый нам фрагмент. Клонирование определяется как процесс выделения заданной последовательности ДНК и получения многих её копий с использованием организмов (здесь репликация). Основной подход предполагает использование бысто делящихся организмов (чаще всего бактериальных клеток, обычно E.coli) для размножения введенной в них чужеродной ДНК. В нашем разделе о клонировании ДНК рассмотрим клонирование с использованием клеток бактерий E. coli, а также стратегию ПЦР-клонирования (клонирование ПЦР-амплифицированных продуктов). Процесс самой ПЦР (полимеразной цепной реакции), как метод амплификаци нуклеиновых кислот in vitro рассмотрим отдельно (Прим.: ПЦР можно рассмотреть и вначале - важно понимать, что получение фрагмента ДНК (гена) с помощью ПЦР возможно, только если мы знаем его нуклеотидную последовательность или хотя бы последовательность его начала и окончания, для того, чтобы можно было синтезировать праймеры).
BAC-КЛОНИРОВАНИЕ
Искусственная бактериальная хромосома (bacterial artificial chromosome, BAC) — векторная система на основе F-плазмиды E. coli, используемая для клонирования длинных (150—350 тыс. п.н.) последовательностей ДНК. Плазмида кодирует гены, регулирующие репликацию и контролирующие копийность (1—2 молекулы на клетку). Искусственные бактериальные хромосомы часто используются для секвенирования геномов организмов в различных проектах, например в проекте Геном человека. Короткий фрагмент ДНК исследуемого организма вставляется в хромосому, а затем амплифицируется и секвенируется. После этого прочитанные последовательности выравниваются in silico в результате чего получается полная последовательность генома организма. Сейчас такой подход был вытеснен более быстрыми и менее трудоёмкими методами секвенирования, например методом дробовика или методами секвенирования нового поколения.
На рисунке - этапы BAC-клонирования фрагмента ДНК с использованием вектора (плазмиды), содержащего ген lac Z (изображены этапы до выделения плазмид с клонированным фрагментом (рис. 22)).

Рис. 21. Этапы клонирования фрагмента ДНК с ипользованием кишечной палочки и вектора, содержащего ген lac Z (все этапы см. ниже) Ген lac Z, кодирует бета-галактозидазу E.coli. Если вектор, содержащий такой ген, ввести в клетку E.coli с поврежденным собственным геном lac Z, то такие рекомбинантные клетки приобретут способность гидролизовать аналог лактозы (X-Gal) и превращать его в голубой 5-бром-4-хлориндол. Исходные мутантные клетки, не содержащие b-галактозидазу, не способны к этому превращению. Следовательно, на среде с X-Gal исходные нерекомбинантные клетки будут давать белые колонии, а рекомбинантные клетки - голубые.
Процесс клонирования ДНК включает следующие этапы:
- Получение целевых фрагментов ДНК (в том числе генов или их частей с помощью ферментов рестрикции);
- Выбор вектора (Вектор - молекула ДНК или РНК, способная переносить включенные в нее чужеродные гены в клетку, где эти молекулы реплицируются автономно или после интеграции с геномом (хромосомой). Не каждая молекула ДНК или РНК может быть вектором. Существует два основных типа векторов: бактериальные плазмиды и бактериофаги)
- Рекомбинация фрагментов (создание рекомбинантной ДНК –> перераспределение генетического материала ДНК путём разрыва и соединения разных молекул, приводящее к появлению новых комбинаций генов или других нуклеотидных последовательностей).
- Вставка фрагмента ДНК в вектор;
- Введение вектора в популяцию восприимчивых клеток хозяина и трансформация с помощью вектора организма хозяина (т.е. поглощение бактериальной клеткой молекулы ДНК из внешней среды);
- Отбор успешно трансформированной клетки (обычно отбор проводят по генетическим маркерам, которыми помечен вектор. Главным образом маркерами служат гены устойчивости к антибиотикам. Поэтому отбор проводят высевом клеток на среды, содержащие конкретный антибиотик. После высева на этих средах вырастают только клетки, в составе которых находится вектор с генами антибиотиковой устойчивости);
- Размножение отобранной клетки
- Выделение векторных молекул из клетки
- Выделение целевого фрагмента ДНК.
Изображение этапов клонирования

Рис. 22. Схема клонирования участка ДНК (гена) в бактериях
Поскольку при каждом клеточном делении бактерии (как и другие клетки) удваивают свою ДНК, это можно использовать для умножения количества необходимой нам ДНК. Для того, чтобы внедрить наш фрагмент ДНК в бактерию, необходимо «вшить» его в специальный вектор, в качестве которого обычно используют бактериальную плазмиду (небольшую относительно бактериальной хромосомы - кольцевую молекулу ДНК, реплицирующуюся отдельно от хромосомы). У бактерий «дикого типа» часто встречаются подобные структуры: они часто переносятся «горизонтально» между разными штаммами или даже видами бактерий. Чаще всего в них содержатся гены устойчивости к антибиотикам (именно из-за этого свойства их и открыли) или бактериофагам, а также гены, позволяющие клетке использовать более разнообразный субстрат. (Иногда же они «эгоистичны» и не несут никаких функций) Именно такие плазмиды обычно и используют в молекулярно-генетических исследованиях. В плазмидах обязательно содержится точка начала репликации (последовательность, с которой начинается репликация молекулы), целевая последовательность рестриктазы и ген, позволяющий отобрать те клетки, которые обладают этой плазмидой (обычно, это гены устойчивости к какому-нибудь антибиотику).
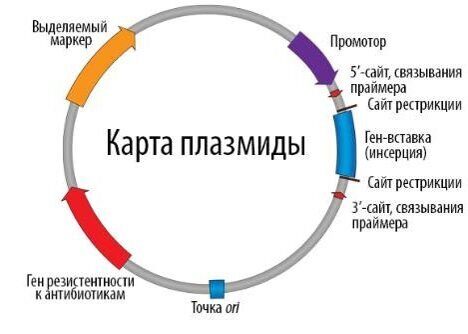
Плазмидная карта может быть прочитана путем понимания ее особенностей, таких как название и размер плазмиды, тип элементов в плазмиде и их относительное положение, а также ориентация промотора.
В плазмиду с помощью рестриктаз и лигаз встраивают необходимый фрагмент ДНК, после чего добавляют ее в культуру бактерий при специальных условиях, обеспечивающих трансформацию – процесс активного захвата бактерией ДНК из внешней среды (риc. 22.B). После этого проводят отбор бактерий, трансформация которых прошла успешно, добавляя соответствующий гену в плазмиде антибиотик: в живых остаются только клетки, несущие ген устойчивости (а, следовательно, и плазмиду). Далее, после роста культуры клеток, из нее выделяют плазмиды, а из них с помощью рестриктаз выделяют «наш» фрагмент ДНК (или используют плазмиду целиком). Если же ген вставили в плазмиду для того, чтобы получить его белковый продукт, необходимо обеспечить культуре условия для роста, а потом просто выделить требуемый белок.
На этом месте сразу же должен возникать вопрос: как же все это возможно было использовать до того, как были расшифрованы геномы, да и чтение последовательности ДНК было еще дорогим и малораспространенным? Положим, с помощью рестрикции и клонирования полученных фрагментов мы получим библиотеку ДНК, то есть набор бактерий, несущих различные плазмиды, содержащие суммарно весь геном (или заметную его часть). Но каким образом мы сможем понять, в каком из фрагментов содержится необходимый ген? Для этого использовали метод гибридизации. Сначала необходимо было выделить белок нужного гена. После чего отсеквенировать его фрагмент, обратить генетический код и получить последовательность нуклеотидов. В соответствии с ней химически синтезировали короткую молекулу ДНК которую, и использовали в качестве зонда для гибридизации.
Стратегии ПЦР-клонирования
 Рис. 24. Клонирование продуктов ПЦР. Схематичный рабочий процесс субклонирования ПЦР-продуктов в сочетании с рестрикционным расщеплением (RE = рестрикционный ферментативный сайт).
Рис. 24. Клонирование продуктов ПЦР. Схематичный рабочий процесс субклонирования ПЦР-продуктов в сочетании с рестрикционным расщеплением (RE = рестрикционный ферментативный сайт).
Клонирование ПЦР-продуктов (ампликонов)
Традиционное клонирование основано на методах рекомбинантных ДНК, которые начинаются с подготовки вектора для получения ДНК-вставки путем расщепления каждого из них ферментами рестрикции. Затем расщепленные фрагменты сращиваются вместе ферментом, называемым лигаза, в процессе, известном как лигирование, с образованием нового вектора, способного экспрессировать интересующий ген. Это может быть самый простой и самый старый метод традиционного клонирования, который заложил основу для исследователей в разработке новых методов клонирования, таких как TA-клонирование, клонирование TOPO® (два из самых простых методов ПЦР-клонирования - ред.), независимое от лигирования клонирование и сборка генов, которые используют уникальные характеристики других модифицирующих ферментов.
ПЦР-клонирование - это метод, при котором двухцепочечные фрагменты ДНК, амплифицированные с помощью полимеразной цепной реакции (ПЦР), лигируются непосредственно в вектор. ПЦР-клонирование предлагает некоторые преимущества по сравнению с традиционным клонированием, которое основано на расщеплении двухцепочечных вставок ДНК с помощью ферментов рестрикции (рестриктазами) для создания совместимых концов, очистки и выделения достаточных количеств и лигирования в аналогично обработанный вектор.

ПЦР-клонирование - это метод клонирования, который значительно сокращает время и усилия, затрачиваемые на реакцию клонирования. Процедура клонирования ПЦР состоит из четырех следующих этапов: (1) Получение фрагмента гена с помощью ПЦР, (2) расщепление геномной ДНК, (3) лигирование в плазмидный вектор и (4) трансформация в бактерии, а затем бактерии будут реплицировать плазмиду.
При ПЦР-амплификации этот метод клонирования требует гораздо меньше исходных шаблонных материалов, которые включают кДНК, геномную ДНК или другую плазмиду, несущую вставку. Кроме того, клонирование ПЦР обеспечивает более простой рабочий процесс, обходя требование о наличии подходящих сайтов рестрикции и их совместимости между вектором и вставкой. Тем не менее, существует ряд соображений, касающихся: праймеров для ПЦР и условий амплификации, выбора метода клонирования и используемых векторов клонирования и, наконец, подтверждения успешного клонирования и трансформации.
Что касается ПЦР-амплификации интересующей последовательности, необходимо разработать праймеры и оптимизировать условия ПЦР (компоненты и циклы) для эффективной и специфической амплификации матрицы (шаблона). Инструменты для конструирования праймеров доступны для биоинформационной оценки и выбора подходящих специфичных для мишени (целевых) последовательностей праймеров для амплификации.
Лигирование требует, чтобы либо вставка, либо вектор имели 5'-фосфорилированные концы; следовательно, если в клонирующем векторе отсутствуют 5'-фосфорилированные концы, к ПЦР-праймерам во время синтеза должны быть добавлены 5' - фосфатные группы или Т4-полинуклеотидкиназы для успешного лигирования (Т4-полинуклеотидкиназа катализирует перенос гамма-фосфата от АТФ на дефосфореллированные 5'-концы ДНК или РНК (5’-концевую ОН-группу). Для оптимизации ПЦР важны также концентрации компонентов реакции, температуры отжига и количество шаблонов.
ТА-клонирование и клонирование по тупым концам

Рис. 23. Общие стратегии ПЦР-клонирования
ТА-клонирование и клонирование по тупым концам представляют собой два самых простых метода ПЦР-клонирования. Их выбор зависит от природы вектора и типа ПЦР-ферментов, используемых при клонировании.
TA Клонирование (также известное как быстрое клонирование) - метод субклонирования который избегает помощи ферментов рестрикции (рестрикционных эндонуклеаз или рестриктаз) и легче и быстрее чем традиционное субклонирование. Этот метод основан на способности тимина (Т) и аденина (А) (комплементарных базовых пар) на различных фрагментах ДНК к гибридизации и, в присутствии лигазы, связываться вместе. Продукты ПЦР обычно амплифицируются с использованием Taq ДНК-полимеразы. Процедура использует терминальную трансферазную активность некоторых термофильных ДНК-полимераз, включая полимеразу Thermus aquaticus (Taq). Taq-полимераза обладает шаблонно-независимой активностью (не обладает корректорской активностью 3'→5'), которая предпочтительно добавляет один дезоксиаденин (3'dA) к 3'-концам двухцепочечной молекулы ДНК, и таким образом большинство молекул ПЦР, амплифицированных Taq-полимеразой, обладают одиночными 3'dT-концами. Полученные ПЦР-продукты с 3'dA-концами легко клонируются в линеаризованный вектор клонирования TA («Т-вектор»), содержащий комплементарные 3' дезокситиминовые (3'dT) концы (Рис. 24).
Технология клонирования TA значительно упрощает традиционное клонирование с помощью одноэтапной стратегии клонирования, которая устраняет необходимость в каких-либо ферментативных модификациях продукта ПЦР и не требует использования праймеров, содержащих сайты ферментов рестрикции. Однако несмотря на относительную простоту, ограничения этого метода включают длину вставки (до 5 кб, здесь кб - килобаза, 1 кб = 1000 пар нуклеотидов, п.н.), невозможность клонирования вставок по направлению и высокую частоту ошибок, связанную с Taq ДНК-полимеразой.
Клонирование по тупым концам (клонирование тупого конца) включает в себя лигирование вставки в линеаризованный вектор, где оба фрагмента ДНК не имеют выступов. Тупые концевые вставки могут быть получены с использованием высокоточных ДНК-полимераз с 3'→5 ' экзонуклеазной или коррекционной активностью. Их корректорская активность улучшает точность последовательности амплифицированных продуктов. Так, например, в отличие от Taq ДНК-полимеразы, Pfu ДНК-полимераза обладает 3'-->5' экзонуклеазной коррекционной активностью, что означает, что по мере сборки ДНК с 5' конца до 3' конца, экзонуклеазная активность немедленно удаляет нуклеотиды, неправильно корорпорированные на 3' конце растущей нити ДНК. Следовательно, фрагменты ПЦР, генерируемые Pfu ДНК-полимеразой, будут иметь меньше ошибок, чем ПЦР вставки, генерируемые Taq-полимеразой. Однако здесь есть и ограничения, которые включают более низкую эффективность лигирования при вставке в тупые векторы клонирования и невозможность клонирования в прямом направлении. Эффективность лигирования может быть повышена путем инкубации ампликонов с Taq ДНК-полимеразой и dATP в процедуре, называемой "3 'dA tailing" (инкубация 20-30 минут при 72°C), а затем очистки продуктов с 3' dA-хвостом (Рис.24).
Технология, стоящая за клонированием TOPO®
Для дальнейшего упрощения и оптимизации рабочего процесса клонирования были разработаны специализированные векторы для размещения вставки в вектор, например, без использования лигазы.
Для примера: Один из таких классов векторов включает в себя векторы клонирования Invitrogen TOPO (одного из брендов известной биотехнологической корпорации Thermo Fisher Scientific), которые содержат ковалентно связанную ДНК-топоизомеразу I, которая функционирует как фермент рестрикции, так и лигазу.
По сравнению с обычными векторами клонирования ПЦР, эти векторы приводят к более короткому времени реакции лигирования (например, 5 минут) и большей эффективности клонирования (например, >95% положительных клонов) и с гораздо более простым протоколом. Кроме того, направленное клонирование продуктов ПЦР может быть достигнуто с помощью специально разработанного ТОПО-вектора с использованием конкретной конструкции праймера - направленное клонирование вектора позволяет клонировать тупые продукты ПЦР в ориентации 5→3 непосредственно в вектор экспрессии с использованием 5-минутной реакции лигирования, тем самым устраняя этапы субклонирования и экономя ваше время.
Рис. 24. Клонирование TOPO® продуктов ПЦР
Ключом к клонированию TOPO® (далее ТОПО-клонированию) является фермент ДНК-топоизомераза I, который функционирует как фермент рестрикции и как лигаза. Его биологическая роль заключается в расщеплении и воссоединении ДНК во время репликации. Топоизомераза вируса осповакцины I специфически распознает пентамерную последовательность 5´-(C/T)CCTT-3´ и образует ковалентную связь с фосфатной группой, присоединенной к 3-тимидину. Фермент расщепляет одну нить ДНК, позволяя ДНК разматываться. Затем он отводит концы расщепленной нити и высвобождается из ДНК. Для того чтобы обуздать религирующую активность топоизомеразы, ТОПО-векторы линеаризуют с помощью топоизомеразы I, ковалентно связанной с каждым 3´ фосфатом. Это позволяет векторам легко связывать последовательности ДНК с совместимыми концами (Рис.24-1,2,3). Лигирование завершается всего за 5 минут при комнатной температуре.
если видео не отображается см. по ссылке →
Независимо от выбора метода клонирования, эффективность клонирования значительно повышается за счет очистки ампликонов ПЦР перед реакцией лигирования. Очистка ПЦР помогает удалить соли, нуклеотиды, неспецифические ампликоны и праймеры-димеры. После лигирования и превращения в соответствующие компетентные клетки полученные колонии необходимо тщательно проверить на наличие правильной вставки, а также ее правильную структуру и ориентацию для последующих исследований с целью анализа слияния генов и/или экспрессии белка.
Основы библиотечного строительства

В молекулярном клонировании построение библиотеки ДНК относится к созданию клонов, которые несут фрагменты ДНК, представляющие полную геномную ДНК (гДНК или англ. gDNA-вида), или комплементарную ДНК (кДНК или англ. cDNA) РНК-транскриптов, представляющих экспрессируемый геном. Путем построения библиотек ДНК, тысячи генетических фрагментов могут быть удобно архивированы и расширены для последующих применений, таких как генотипирование и фенотипический скрининг. Библиотеки гДНК служат полезными инструментами для изучения генетического состава различных видов или мутаций генов, возникающих при таких заболеваниях, как рак. С другой стороны, библиотеки кДНК, с другой стороны, полезны для анализа экспрессии генов и вариантов транскриптов, основанных на типе клетки и происхождении ткани (пространственном), а также временных точках (темпоральном).
Создание библиотек гДНК и кДНК имеет много общих черт, но также и некоторые важные различия. Обе стратегии включают извлечение нуклеиновых кислот, подготовку образцов (например, рестрикционное расщепление), клонирование вектора, введение вектора в подходящего хозяина (например, трансформацию или трансдукцию) и клональный отбор. Поскольку исходные материалы различны между библиотекой гДНК и библиотекой кДНК, их выделение и подготовка используют различные подходы; однако после того, как фрагменты гДНК или кДНК будут клонированы в нужный вектор, можно следовать тому же рабочему процессу.
Для подготовки геномной библиотеки, гДНК извлекается из организма, тканей или клеток, представляющих интерес. Извлеченную гДНК затем расщепляют, выделяют и лигируют в представляющий интерес вектор с совместимыми концами. Частичное расщепление генома часто осуществляется с помощью фермента рестрикции с преобладанием режущих участков, чтобы обеспечить перекрытие последовательности между фрагментами для картирования клонированных вставок (Рис. 25.1).

Рисунок 25.1. Принципиальная схема полного и частичного расщепление фрагмента рестрикционным ферментом с четырьмя режущими участками. Частичное расщепление приводит к наложению последовательностей между фрагментами для отображения. (Для простоты здесь показаны только некоторые возможные частично расщепленные фрагменты.)
Выбор вектора для библиотек гДНК является важным фактором, поскольку фрагменты генов, используемые в конструкциях библиотек, часто бывают большими (например, > 20 kb). Выбор вектора клонирования, в свою очередь, определяет способ доставки векторов, несущих вставку, в хозяина (таблица 1)
Таблица 1. Общие типы векторов, длины клонированных фрагментов и методы доставки векторов при создании библиотеки.
|
Тип вектора
|
кДНК (kb)
|
Способ доставки вектора
|
|
Плазмида
|
20
|
Трансформация
|
|
фаг λ
|
25
|
Трансдукция
|
|
Космид
|
45
|
Трансдукция
|
|
Фаг P1
|
100
|
Трансдукция
|
|
BAC (бактериальная искусственная хромосома)
|
300
|
Электропорация
|
|
YAC (дрожжевая искусственная хромосома)
|
1,000
|
Трансформация (дрожжи)
|
Продукты лигирования или рекомбинантную ДНК можно вводить непосредственно в бактериальные клетки путем трансформации или упаковывать в бактериофаг для инфицирования или “трансдукции” клеток-хозяев (рис. 25.2). Преобразованные или трансдуцированные клетки предназначены для последующего архивирования, расширения и секвенирования в последующих экспериментах. Последовательности всего генома многих организмов, включая первую последовательность всего человеческого генома, были определены с использованием этой базовой стратегии в начале 2000 года.

Рис. 25.2. Схематичный рабочий процесс подготовки геномной библиотеки с использованием фагового вектора λ. Образец геномной ДНК частично расщепляется с помощью Sau3AI, после чего выделяются ~20-килобайтные фрагменты (идеальный размер для вирусной упаковки) для лигирования с фрагментами вирусного гена.
Для подготовки библиотеки кДНК общая РНК извлекается из биологического источника (например, клетки, ткани и т.д.), после чего мРНК обратно транскрибируется в комплементарную ДНК (кДНК). Этот процесс известен как синтез первой цепи кДНК. Затем синтезируется вторая нить для получения двухцепочечных кДНК. Получающиеся в результате двухцепочечные фрагменты могут быть лигированы непосредственно в вектор клонирования с тупым концом (случайное клонирование) или «помечены» на концах сайтами рестрикции для направленного клонирования (рис. 25.3).
Библиотеки кДНК, которые обеспечивают хорошее и точное представление о экспрессированном геноме, зависят от нескольких факторов, включая качество и целостность исходной популяции мРНК. Для этапов обратной транскрипции также важно, чтобы обратная транскриптаза была способна синтезировать кДНК из смешанной и сложной популяции, включая длинные матрицы РНК и редкие транскрипты РНК, для адекватного охвата в библиотеках. Используя базовую стратегию, представленную на рисунке, многие препараты библиотеки кДНК были использованы для создания комплексных коллекций, включая коллекцию генов млекопитающих (MGC), крупнейшую спонсируемую NIH публичную коллекцию библиотек клонов кДНК видов млекопитающих, включая человека, мышь и крысу.

Рис. 25.3. Стратегии клонирования кДНК с использованием мРНК с поли(А)-хвостом (остатками аденозинмонофосфата). При случайном (ненаправленном) клонировании двухцепочечные кДНК связываются непосредственно с тупым концом вектора клонирования. При направленном клонировании адаптеры с редкими сайтами рестрикции (например, NotI и SalI) лигируют с двухцепочечными концами кДНК для клонирования в вектор с совместимыми концами.
Способ клонирования и секвенирования Дробовика

Метод ШОТГАН (Shotgun) или Метод Дробовика
Метод дробовика или метод "Шотган" (от англ. shotgun) - это метод, используемый для секвенирования (определения нуклеотидной последовательности) длинных участков ДНК. Суть метода состоит в получении случайной массированной выборки клонированных фрагментов ДНК интересующего организма, на основе которых может быть восстановлена исходная последовательность ДНК.
После построения библиотеки одна из целей состоит в том, чтобы охарактеризовать клоны путем секвенирования вставок. Размеры вставок, представленных в этих библиотеках, часто могут варьироваться от 25 до 300 Кб (килобаз), в зависимости от типа векторов и размера генома интересующего организма. Для секвенирования по Сэнгеру, когда-то самого распространенного метода секвенирования ДНК, верхний предел реакции секвенирования с хорошим качеством считывания обычно составляет менее 1 Кб (<1000 пар нуклеотидов, п.н.). Чтобы преодолеть эту дилемму, исследователи могут обратиться к клонированию дробовика и секвенированию. В этом подходе большие клонированные вставки далее фрагментируются физическими или ферментативными средствами и субклонируются в другой вектор; более мелкие клонированные фрагменты затем секвенируются. После этого эти последовательности повторно собираются на основе перекрытий последовательностей (называемых контигами) с использованием программ биоинформатики, чтобы в конечном итоге получить исходную длинную последовательность (рис. 25.4).

Рисунок 25.4. Схематичный рабочий процесс клонирования дробовика.
Секвенирование дробовика играет важную роль в секвенировании всего генома многих организмов, начиная от вирусов и бактерий до человека. Этот метод может быть использован для секвенирования генома de novo, а также для улучшения качества уже секвенированного генома путем проверки считывания и заполнения пробелов. Во время первого секвенирования человеческого генома финансируемый проект «Геном человека» («Human Genome Project») использовал дробовое секвенирование больших фрагментов генов, которые были клонированы в бактериальную искусственную хромосому (или BAC-вектор от bacterial artificial chromosome – см. табл.1.). Геномные позиции клонированных фрагментов были определены до клонирования дробовика, что облегчило сборку их последовательности дробовика. Следовательно, этот метод известен как иерархическое секвенирование дробовика (рис.25.5-А). Он также называется секвенированием clone-by-clone («клон за клоном») из-за использования клонов BAC в качестве источника.
Параллельно с проектом «Геном человека» другой финансируемый частным образом проект секвенирования всего генома, возглавляемый Крейгом Вентером (John Craig Venter), использовал стратегии секвенирования дробовика непосредственно на ДНК генома человека (вместо клонированных фрагментов, которые уже были нанесены на карту). Этот процесс известен как цельногеномный подход дробовика (рис.25.5-B). В теории секвенирование дробовика не требует предварительной информации о геноме или генетических картах и сэкономит время и ресурсы. Тем не менее, полезно иметь справочные генетические карты при сборке последовательностей, поскольку для полногеномного дробового подхода требуется большое количество вычислительной мощности, особенно для организмов с большими геномами.

Рисунок 25.5. Схематичный рабочий процесс двух подходов к секвенированию дробовика, используемых при секвенировании всего генома человека.
- Иными словами, при секвенировании с помощью дробовика всего генома весь геном случайным образом разрезается на мелкие фрагменты (соответствующие размеру для секвенирования), а затем вновь собирается. См. здесь →
- В иерархическом секвенировании дробовика геном сначала разбивается на более крупные сегменты. После определения порядка этих сегментов, они разрезаются на фрагменты, соответствующие размеру для секвенирования. См. здесь →

Таким образом, процесс секвенирования методом дробовика (или секвенирования дробовиком) состоит из нескольких этапов: Сначала секвенируемая ДНК подвергается амплификации (т.е. увеличению числа ее копий). Полученные копии ДНК разрезаются на фрагменты с помощью сайт-неспецифичных нуклеаз. Сайт-неспецифичность важна для того, чтобы получались перекрывающиеся фрагменты. Как уже описывалось выше, из полученных фрагментов строится геномная BAC библиотека путём встраивания фрагментов в некоторый вектор.
 Из полученной геномной библиотеки случайным образом выбирается некоторое подмножество фрагментов, каждый из которых секвенируется, например, методом Сэнгера. Затем с помощью специального программного обеспечения из полученных нуклеотидных последовательностей фрагментов, называемых ридами, собирается нуклеотидная последовательность исходной ДНК.
Из полученной геномной библиотеки случайным образом выбирается некоторое подмножество фрагментов, каждый из которых секвенируется, например, методом Сэнгера. Затем с помощью специального программного обеспечения из полученных нуклеотидных последовательностей фрагментов, называемых ридами, собирается нуклеотидная последовательность исходной ДНК.

См. дополнительное видео по теме: Алгоритмы сборки
Геномные библиотеки и картирование генома
(продолжение темы с некоторыми дополнениями)
Даже такие небольшие геномы, как у фагов или вирусов, оказываются слишком велики для прямого секвенирования. Для изучения структуры генома его расщепляют на мелкие фрагменты, определяют нуклеотидную последовательность фрагментов путем секвенирования, а затем с помощью компьютерных методов создают полную ДНК- или РНК-карту генома. Генетический код избыточен, поэтому для изучения структуры генома (генетического картирования) необходимо иметь соответствующие маркерные участки (маркеры), как правило, это – ДНК-маркирующие сайты STS (sequence-tagged sites).
ГЕНОМНАЯ БИБЛИОТЕКА. Геномная библиотека – это множество клонированных фрагментов ДНК, в совокупности охватывающих весь геном. Для получения библиотеки геном расщепляют на фрагменты, а затем полученные фрагменты встраивают в векторы. Для облегчения процедуры упорядочивания фрагменты должны быть достаточно крупными, поэтому для ферментативного расщепления используют такие рестриктазы, сайты которых встречаются в геноме относительно редко. Примером может служить рестриктаза Not I, узнающая последовательность 5'-GCGGCGC-3. Необходимо учитывать, что частота встречаемости той или иной последовательности в геноме зависит от GC состава ДНК и от частоты повторяемости участков ДНК.
ВЕКТОРЫ. При создании геномной библиотеки фрагменты ДНК встраивают в векторы, а затем полученные ДНК-конструкции вводят в клетки. В таком виде фрагменты ДНК доступны для выделения, анализа и амплификации. Чем крупнее геном, тем большим количеством клонов представ лена полная геномная библиотека. Для больших эукариотических геномов наиболее подходящими векторами являются космиды или дрожжевые/бактериальные искусственные хромосомы YAC/BAC (yeast/bacterial artificial chromosomes). В эти векторы можно встраивать фрагменты ДНК размером до 2 млн п.н., однако для прямого секвенирования такие фрагменты слишком велики. Субклонирование осуществляют, используя векторы на основе генома фага λ, например космиды, позволяющие встраивать фрагменты ДНК размером 30–45 т.п.н.
КАРТИРОВАНИЕ ГЕНОМА. Классический метод картирования генома основан на наблюдении за наследованием сцепленных фенотипических признаков. Так, для представителей аскомицетов Neurospora crassa и дрожжей Saccharomyces cerevisiae можно построить физическую карту генома, анализируя тетрады: уэтих организмов споры в аске располагаются в том порядке, в котором они образовались при мейозе, поэтому изучение распределения фенотипическихпризнаков облегчается. Методы генетической инженерии значительно расширили возможности картирования генома. Результаты секвенирования фрагментов генома, полученных после ферментативного расщепления, позволяют определить полную структуру небольшого генома или выделить уникальные маркерные последовательности больших геномов. С помощью хорошо подобранных праймеров для ПЦР или зондов для гибридизации можно получать специфичные маркеры для больших фрагментов ДНК, на- пример для флуоресцентной гибридизации in situ (FISH – fluorescence in situ hybridization). «Причесывание» ДНК – способ упорядочивания ДНК для маркировки. Для этого в раствор, содержащий крупные фрагменты ДНК (около 10 т.п.н.), погружают обработанный полилизином носитель. При медленном удалении носителя из раствора (со скоростью примерно 0,3 мм/с) ДНК-фрагменты располагаются на нем параллельно друг другу, что значительно облегчает процесс их маркировки. Для анализа больших эукариотических геномов ранее создавали хромосомо-специфичные библиотеки. Их сохраняют посредством окрашивания флуоресцентными красителями и клеточной сортировки, зависящей от интенсивности флуоресценции (FACS), так как количество красителя, связанного с отдельными хромосомами, меняется.
ДНК-МАРКИРУЮЩИЕ САЙТЫ (STS) – это специфические участки ДНК размером 100–500 п.н., которые встречаются в геноме всего один раз. Изначально предполагается, что STS не содержат повторяющихся последовательностей. STS часто получают из больших фрагментов генома, содержащихся в клонотеке (библиотеки BAC или YAC). Если при секвенировании генома удалось получить коллекцию таких ДНК-маркеров, то, создав на их основе праймеры для ПЦР, можно определить их взаиморасположение в геноме: размер продукта ПЦР зависит от расстояния между маркерами в геноме. В банке данных можно найти более крупные последовательности ДНК, несущие два соседних ДНК-маркера. ДНК-маркеры представляют собой очень удобный инструмент для анализа сцеп ленных генов.
Геном прокариот
ВВЕДЕНИЕ. При создании генетических карт прокариот анализируют изменения фенотипических признаков, происходящие в результате процессов конъюгации (перенос ДНК из одной контактирующей клетки в другую), трансдукции (передача ДНК между двумя бактериальными клетками с помощью фага) или трансформации (введение очищенной ДНК в клетку). Первая физическая карта генома (полная нуклеотидная последовательность) была получена в 1995 г. Для построения карты генома используют метод выстраивания клонированных фрагментов в контиги и метод «дробовика» (shotgun).
ГЕНЕТИЧЕСКИЕ КАРТЫ. За изменением многих фенотипических признаков можно наблюдать при размножении бактерий. В качестве маркеров можно использовать способность бактерии образовывать жгутики или споры, а также устойчивость к антибиотику. Для изучения путей биосинтеза используют мутантные штаммы, в которых блокирована одна из стадий биосинтеза. Для роста таких мутантов необходимо наличие в среде веществ, которые бактерия сама больше не может синтезировать. Немаловажным преимуществом бактерий является короткий жизненный цикл (время между образованием клетки и ее делением), часто он занимает менее 1 ч. Сопоставление фенотипических изменений и времени проникновения тех или иных участков ДНК в клетку-реципиент при конъюгации позволяет определить взаиморасположение генов, которые кодируют эти признаки. Таким образом, карты генома прокариот строят в масштабе минут или центисом. Процесс переноса всего генома E. coli в клетку-реципиент при 37 °С занимает 100 мин.
ФИЗИЧЕСКОЕ КАРТИРОВАНИЕ: ПОЛУЧЕНИЕ КОНТИГОВ. Важной задачей является идентификацияклонов геномной библиотеки, содержащих перекрывающиеся фрагменты ДНК (контиги) (от англ. contiguous– непрерывный). Исходя из этой информации,можно реконструировать непрерывный ряд клонированных фрагментов, составляющих весь геном. Дляпоиска контигов применяют метод генетической«дактилоскопии» (clone fingerprinting), например,сравнивая рестриктные карты или перекрывающиесяSTS и выявляя «соседние» клоны. Полезную информацию можно получать из анализа распределениягенетических маркеров. Позиционное клонирование(т. е. поиск гена, соседствующего с маркером)осуществляют методом «прогулки по хромосоме»(chromosome walking). Уникальный участок ДНК,примыкающий к одному из концов клонированногофрагмента, используют в качестве зонда для гибридизации с геномной библиотекой. Некоторые гибридизующиеся клоны полностью перекрываются с исходным участком, а другие включают новые сегменты. Таким образом карта расширяется. При повторении этой процедуры совершается «прогулка» на большее расстояние.
ФИЗИЧЕСКОЕ КАРТИРОВАНИЕ ГЕНОМА: МЕТОД «ДРОБОВИКА» (SHOTGUN). Физическая карта геномапредставляет собой его полную нуклеотидную последовательность. В настоящее время возможно секвенирование фрагментов ДНК размером до 600 п.н.Для создания физической карты геном разрезают нанебольшие фрагменты с помощью рестриктаз или ультразвука, затем секвенируют клонированные фрагменты и с помощью компьютерных программ устанавливают последовательность их расположения в геноме. Современные компьютерные технологии позволяют быстро составлять кар ты и анализировать бактериальные геномы (размером, как правило, до 5 млн п.н.). Наличие генетических карт и постоянное пополнение данных о взаиморасположении генетических маркеров позволяет проводить корреляцию между физической и генетической картами. Так, к 1990 г. на хромосоме E. coli были локализованы более 1400 генетических маркеров, расположенных в среднем через каждые 3300 п.н. (размер всего генома E. coli составляет 4,64 млн п.н.).
БИОИНФОРМАТИКА. Большинство компьютерных программ для анализа генома основываются на поиске гомологичных последовательностей, поэтому даже такое редкое явление, как ошибка секвенирования (стандартная точность секвенирования составляет 99%), приводит к появлению неточностей практически во всех последовательностях. По этой причине в компьютерные банки данных вносят информацию, подтвержденную многократным секвенированием.
5. ПОЛИМЕРАЗНАЯ ЦЕПНАЯ РЕАКЦИЯ (ПЦР)
Полимеразная цепная реакция (ПЦР) — метод молекулярной биологии, позволяющий добиться значительного увеличения малых концентраций определённых фрагментов нуклеиновой кислоты (ДНК или РНК) в биологическом материале (пробе). Помимо амплификации ДНК, ПЦР позволяет производить множество других манипуляций с нуклеиновыми кислотами (введение мутаций, сращивание фрагментов ДНК) и широко используется в биологической и медицинской практике.
амплификация ДНК-фрагментов

Полимеразная цепная реакция — молекулярно-биологический метод, позволяющий добиться колоссального (до 1012 раз) увеличения числа копий определенного фрагмента ДНК in vitro. Она была изобретена Кэри Муллисом (Kary Mullis) в 1983 году, за что в 1993 году он получил Нобелевскую премию по химии (совместно с М. Смитом).
Давайте вначале попытаемся извлечь максимум информации из самого названия метода. Ааббревиатура ПЦР (произносим «пэ-цэ-эр») обозначает «Полимеразная Цепная Реакция». Разберем эти слова по порядку. Слово «полимеразная» говорит нам о полимеризации, то есть построении полимера из мономеров. Слово «цепная» означает зависимость следующих этапов реакции от продуктов предыдущих. Наконец, слово «реакция» предполагает превращение одних веществ в другие, отличающиеся от исходных по химическому составу и (или) строению. Теперь посмотрим, одно из определений термина:
Полимеразная цепная реакция (ПЦР) - экспериментальный молекулярно-биологический метод, который основан на катализируемой ДНК-полимеразой реакции, и который позволяет амплифицировать (от английского amplify - многократно увеличивать, усиливать) in vitro малые концентрации определённых фрагментов ДНК в биологическом материале в миллионы раз в течение нескольких часов.
Копирование ДНК при ПЦР осуществляется специальным ферментом - ДНК-полимеразой, как и в клетках живых организмов. ДНК-полимераза, двигаясь по одиночной цепи ДНК (матрице), синтезирует комплементарную ей последовательность ДНК. Важно, что ДНК-полимераза не может начать синтез цепи ДНК «с нуля», ей необходима короткая «затравочная» цепь РНК или ДНК, к которой она может начать присоединять нуклеотиды.
Основной принцип ПЦР состоит в том, что реакция полимеризации (синтеза полимерной цепи ДНК из мономерных нуклеотидных звеньев) инициируется специфическими праймерами (короткими фрагментами «затравочной» ДНК) в каждом из множества повторяющихся циклов. Специфичность ПЦР определяется способностью праймеров «узнавать» строго определенный участок ДНК и связываться с ним согласно принципу молекулярной комплементарности.
В обычной реакции ПЦР используется пара праймеров, которые «ограничивают» амплифицируемый участок с двух сторон, связываясь с противоположными цепями ДНК-матрицы. Для многократного увеличения количества копий исходной ДНК нужна цикличность реакции. Как правило, каждый из последовательно повторяющихся циклов ПЦР состоит из трех этапов:
- денатурации, или «плавления» ДНК, когда двухцепочечная ДНК под действием высокой температуры переходит в одноцепочечное состояние;
- связывания (отжига) праймеров с матричной ДНК;
- элонгации, или удлинения цепи.
Смена этапов каждого цикла осуществляется путем изменения температуры реакционной смеси (рис. 27 а,б,в). Сначала праймеры могут связаться только с определенной последовательностью исходной ДНК, но в последующих циклах они связываются с копиями этой последовательности, синтезированными в предыдущих циклах. При этом количество основного продукта ПЦР (копии последовательности ДНК, ограниченной праймерами) теоретически удваивается в каждом цикле, то есть растет с числом циклов экспоненциально.
Как проводится ПЦР?
ПЦР можно условно разделить на два этапа – подготовительный и основной. Во время первого этапа идет подготовка реакционной смеси, иными словами, в одну пробирку скапываются все необходимые компоненты. Длится этот этап, в зависимости от количества пробирок и способа скапывания, обычно до часа. Во время второго этапа происходит собственно ПЦР, при этом пробирки с реакционной смесью помещают в специальный прибор и оставляют на несколько часов (обычно 3-5). Разберем подробно каждый из этих этапов.
Подготовительный этап. Компоненты для ПЦР.
 Реакционную смесь обычно готовят в специальных тонкостенных пробирках типа Эппендорф объемом 200 мкл (рис. слева). Для этого в пробирки последовательно добавляют все компоненты в рассчитанном объеме для получения необходимой финальной концентрации. Перечислим все необходимые компоненты и их роль в реакции.
Реакционную смесь обычно готовят в специальных тонкостенных пробирках типа Эппендорф объемом 200 мкл (рис. слева). Для этого в пробирки последовательно добавляют все компоненты в рассчитанном объеме для получения необходимой финальной концентрации. Перечислим все необходимые компоненты и их роль в реакции.
Поскольку, как уже упоминалось, ПЦР является модифицированной реакцией репликации, реакция невозможна без ДНК-матрицы – той ДНК, которая содержит необходимый для многократного копирования фрагмент. Матрицей может служить любая молекула ДНК любого живого организма, при этом концентрация может быть очень низкой – всего несколько копий ДНК. Конечно же, для построения новой цепи ДНК необходим фермент ДНК-зависимая ДНК- полимераза. При проведении ПЦР обычно используют особую термостойкую версию фермента – Taq-полимеразу – произносится «так полимеразу». Фермент был получен из термофильной бактерии Thermus aquaticus. Первая буква рода и две первые буквы вида бактерии на латинском языке определили название фермента - Taq.
Ферменту для построения новой цепи необходимы «кирпичики» - предшественники нуклеотидов. Действительно, в реакционную смесь обязательно добавляют дезоксинуклеозиды трифосфаты, которые являются не только источником новых нуклеотидных звеньев в строящейся ДНК, но и источником энергии за счет своих макроэргических связей между остатками фосфатов. За счет этого АТФ в реакционную смесь не добавляют. В естественных условиях в клетке репликация начинается в особых местах – точках начала репликации. При проведении ПЦР начало репликации определяют праймеры. Праймер – относительно короткий (20-40 нуклеотидов) одноцепочечный фрагмент ДНК, комплементарный одной из цепей ДНК-матрицы. Праймер служит затравкой для ДНК-полимеразы подобно РНК-затравке в естественном процессе репликации. Для ПЦР нужно два праймера – прямой (на английском - forward) и обратный (на английском - reverse). Прямой праймер «садится» на транскрибируемую (матричную) цепь в начале фрагмента и обеспечивает синтез дочерней цепи от начала к концу. Обратный праймер соединяется с нетранскрибируемой цепью ДНК в конце фрагмента и обеспечивает синтез другой дочерней цепи в направлении от конца к началу:

Схематическое изображение синтеза комплементарной цепи и расположения мест присоединения праймеров.
Цветным шрифтом (синим и красным) обозначен фрагмент двухцепочечной ДНК, число копий которого необходимо многократно увеличить. Стрелками обозначены направления, в которых будет идти синтез новых цепей. 20-ти нуклеотидные праймеры (прямой и обратный) приведены в зеленом и желтом прямоугольниках (соответственно). Их последовательность строго комплементарна цепям ДНК-матрицы.
Фермент ДНК-полимераза для функциональной активности требует наличие в реакционной среде ионов магния. Поэтому ионы магния, обычно в составе MgCl2, обязательно добавляют в пробирку. ДНК-полимераза, подобно многим другим ферментам требует для оптимальной активности определенного уровня кислотности. Поэтому обязательным компонентом реакционной смеси является специальный ПЦР-буфер, который обеспечивает поддержание оптимального pH. Наконец, последним необходимым компонентом реакции ПЦР является вода. С одной стороны, она является средой для протекания реакций, с другой стороны, меняя ее объем, добиваются одинакового объема реакционной смеси и нужной концентрации всех компонентов. Все компоненты с кратким перечислением их функций приведены в таблице 1.
Таблица 1. Роль компонентов реакционной смеси ПЦР
|
Компоненты реакционной смеси ПЦР
|
Роль в реакции
|
|
|
1.
|
ДНК-матрица
|
Матрица для синтеза дочерних цепей - образец ДНК с целевой последовательностью. Он может быть от любой клетки, содержащей ДНК.
|
|
2.
|
Taq-полимераза
|
Это фермент, который на самом деле синтезирует новые нити ДНК. Обычно используются термостабильные ДНК-полимеразы, такие как Taq, потому, что их активность не нарушается при высоких температурах, а это означает, что вам не нужно добавлять больше полимеразы после высокотемпературных этапов термоцикла.
|
|
3.
|
Дезоксинуклеозиды трифосфаты (dNTPs)
|
Нуклеотиды со свободным основанием, которые будут использоваться для создания новых нитей ДНК.
|
|
4.
|
Два праймера – прямой и обратный
|
Служат затравкой для ДНК-полимеразы. Праймеры - это короткие одиночные цепи ДНК, которые связываются по обе стороны от последовательности-мишени, чтобы обеспечить амплификацию области, которую они охватывают (взгляните на диаграмму). Они обычно около 18-30 нуклеотидов в длину.
Праймеры сконструированы таким образом, что они комплементарны областям, которые фланкируют целевой ген, поэтому они будут связываться с этими последовательностями (и только с этими последовательностями). Они также предназначены для включения определенного количества С и G нуклеотидов для изменения температуры, при которой они будут прикрепляться (отжигать) к ДНК.
|
|
5.
|
Ионы магния (Мg2+)
|
Являются кофакторами для ДНК-полимеразы. Ионы Mg2+ обеспечивает раствор MgCl2. Mg2+ образует комплексы с dNTPs (именно эти комплексы являются субстратом для Taq-полимеразы), праймерами и матрицей ДНК и поэтому концентрация Mg2+ влияет на отжиг праймеров, температуру диссоциации матричной и амплифицированной ДНК.
|
|
6.
|
ПЦР-буфер
|
Позволяет поддерживать оптимальное значение рН раствора для проведения реакции ПЦР. Служит химической средой для поддержания активности и стабильности ДНК полимеразы.
|
|
7.
|
Вода
|
Среда для протекания реакции, доведение объема реакционной смеси до необходимого
|
Основной этап: что происходит в пробирке?
После того как смесь готова, начинается основной этап ПЦР. Основной этап состоит из 20 – 40 циклов. Каждый цикл протекает по одной и той же схеме и состоит из трех стадий – денатурация, отжиг (или гибридизация) праймеров, элонгация. Каждая стадия протекает при строго определенной температуре (рис. 28). Для последовательного создания и поддержания необходимого температурного режима необходимо специальное оборудование – амплификатор (рис. 26). Разберемся подробнее с каждой из стадий.
 Рис. 26. Амплификатор для проведения ПЦР.
Рис. 26. Амплификатор для проведения ПЦР.
Первая стадия называется денатурация или плавление. Во время этой стадии происходит разрушение водородных связей в ДНК-матрице за счет кратковременного нагревания пробирки со смесью до 90-95°С (рис. ?А). При этом ДНК-матрица из двуцепочечной превращается в одноцепочечную, что делает возможным присоединение праймеров к комплементарным участкам. Именно во время стадии денатурации крайне важна термостабильность фермента Taq-полимеразы. Если бы использовалась обычная термочувствительная ДНК-полимераза, то она, будучи по химической природе белком, при столь высокой температуре подвергалась бы необратимой денатурации. Для присоединения праймеров необходимо снизить температуру в среднем до 55-65°С. Температура рассчитывается теоретически в зависимости от длины и нуклеотидного состава праймеров, а потом оптимизируется экспериментально. Процесс присоединения праймеров по краям нужного фрагмента называется отжигом или гибридизацией праймеров (рис. 27 б).
 Рисунок 27 (слева). Схематическое изображение трех стадий одного цикла ПЦР (См. также рис. 1 и рис. 2.)
Рисунок 27 (слева). Схематическое изображение трех стадий одного цикла ПЦР (См. также рис. 1 и рис. 2.)
Прямоугольниками выделены фрагменты ДНК, которые необходимо амплифицировать. Точками обозначены водородные связи между комплементарными участками. Стрелками обозначены праймеры и направления синтеза новых цепей ДНК. Штриховой линией обозначены цепи, которые синтезированы на этапе элонгации. Краткое описание всех этапов и условия их протекания приведены в таблице ?.
Подбор оптимальной температуры для отжига праймеров очень важен. Если температура ниже оптимальной, то праймеры будут присоединяться в местах, которые не полностью им комплементарны, что приведет к амплификации неспецифических участков ДНК. В случае более высокой температуры праймеры, наоборот, с трудом будут присоединяться к ДНК-матрице или не будут присоединяться вовсе.
После того, как праймеры присоединились к комплементарным участкам в начале и конце фрагмента, температуру поднимают до 72°С, поскольку эта температура оптимальна для функционирования Taq-полимеразы. Фермент начинает удлинять праймеры в обоих направлениях. Этот этап называется элонгацией (рис. 27в). Длительность его определяется длиной фрагмента, который необходимо амплифицировать. На каждую тысячу пар нуклеотидов отводится в среднем 1 минута. Краткое описание всех этапов и условия их протекания приведены на рис. 28 и в табл. 2.

Рисунок 28. Этапы ПЦР
Шаг 1: 90-95oC - Это активирует энзим полимеразу; Шаг 2: 90-95oC - Стадия денатурации: высокая температура разделяет две нити ДНК, что позволяет другим агентам в коктейле ПЦР получать доступ к генетической информации; Шаг 3: 55-65oC - Этап отжига: при этой более низкой температуре праймеры могут связываться с областями рядом с целевым геном, который мы хотим амплифицировать. Это связывание имеет решающее значение, так как праймеры выступают в качестве отправной точки для синтеза новых цепей, которые станут копиями целевой области; Шаг 4: около 72oC - Стадия синтеза: это оптимальная температура для работы фермента, позволяющая эффективно включать dNTPs полимеразой, чтобы распространить праймеры в полноразмерные копии целевой области; Повторение шагов 2, 3 и 4 - Повторение этапов разделения, отжига праймера и синтеза позволяет делать больше копий. Количество копий целевой области увеличивается экспоненциально, так как с каждым циклом общее количество копий удваивается, как показано на диаграмме ниже; Шаг 5: около 72°С - следует за повторением шагов 2-4 и гарантирует, что синтез цепей был завершен до того, как реакция остановилась; Шаг 6: 4°С - охлаждает реакцию до температуры, при которой она останавливается.
Таблица 2. Этапы ПЦР и условия их протекания
|
Что происходит
|
Температура
|
Длительность
|
|
Этап 1: Денатурация (плавление) |
||
|
Водородные связи разрываются, двуцепочечные
молекулы ДНК распадаются на одноцепочечные
|
90-95°С
|
15 секунд
|
|
Этап 2: Отжиг (гибридизация) праймеров |
||
|
Водородные связи образуются между комплементарными фрагментами ДНК. Одноцепочечные праймеры гибридизуются с участками ДНК в начале и конце нужного фрагмента
|
55-65°С
|
30 секунд
|
|
Этап 3: Элонгация |
||
|
Taq-полимераза удлиняет праймеры в направлении 5´-3´, синтезируя дочерние цепи
|
72°С
|
60 секунд на тысячу пар оснований
|
Что происходит в течение первых трех циклов ПЦР?

Рис. 29. Первые три цикла полимеразной цепной реакции
Для удобства возьмем изначально одну копию ДНК в качестве матрицы (А) (см. рис. 29). К концу первого цикла нужный фрагмент будет удвоен (Б и В), при этом одна из цепей каждого продукта будет идентична исходной матрице (обозначена серым цветом), а другая будет вновь синтезированная (обозначена черным цветом). Важно отметить, что продукты первого цикла не полностью идентичны исходной матрице, поскольку удваивается не вся ДНК, а только необходимый фрагмент.
Далее продукты первого цикла – Б и В – становятся матрицами для второго цикла. Поскольку они при денатурации распадутся на четыре одноцепочечные молекулы ДНК, каждая из которых станет местом присоединения одного из праймеров, в конце второго цикла образуется четыре продукта – Г, Д, Е и Ж.
В третьем цикле матрицами будут служить продукты второго цикла, которые при денатурации распадутся на 8 одноцепочечных молекул. Это означает, что в конце третьего цикла в пробирке будет 8 двуцепочечных копий необходимого фрагмента. Обратите внимание, что доля исходных цепей (обозначена серым цветом) в конце первого цикла составляет 50%, в конце второго цикла – 25%, а в конце третьего только 12,5%. Также отметьте, что только в конце третьего цикла впервые появляются продукты без одноцепочечных «хвостов» - И и О. С каждым последующим циклом доля таких продуктов будет возрастать, а доля исходных цепей уменьшаться.
А теперь посмотрим на протекание ПЦР с математической точки зрения. Возможно вы уже заметили, что увеличение числа копий ДНК происходит в геометрической прогрессии. При этом количество копий нужного фрагмента ДНК после проведения ПЦР в идеальных условиях можно описать следующей формулой:
N = Ax2b, где
N – количество копий ДНК в конце реакции, A – исходное количество копий ДНК, b – количество циклов ПЦР. На практике, количество продуктов будет несколько ниже идеально рассчитанного, однако эта разница будет незначительной и количество продуктов после 20 циклов действительно будет стремиться к миллиону!
Как «увидеть» продукты ПЦР?
Для того, чтобы проверить качество и количество продуктов ПЦР, как правило, используют метод агарозного гель-электрофореза. Для этого достаточно взять около 10 микролитров продуктов ПЦР, внести в лунку агарозного геля и посмотреть на результат разделения в ходе электрофореза.
Усовершенствование технологии ПЦР
Техническое оформление смены температуры реакционной смеси в последнее время стремительно развивалось. Сначала ПЦР осуществлялась при помощи трех водяных бань, настроенных на разную температуру: для денатурации ДНК, отжига праймеров и полимеризации. Пробирки переносились из одной водяной бани в другую «по кругу», благодаря чему происходила смена температуры на разных этапах цикла. Существовали и варианты приборов, где в водяную баню, в которой находились пробирки с реакционной смесью, поочередно подавалсь вода разной температуры. Смена циклов в этих случаях занимала много времени, и процесс плохо поддавался автоматизации.
Для осуществления ПЦР в основном используются приборы (термоциклеры), которые изменяют температуру автоматически на основе заданной программы. В термоциклерах пробирки с реакционной смесью помещаются в металлический блок, температура которого изменяется с помощью элемента Пельтье. Элемент Пельтье позволяет изменять температуру блока с большой скоростью, что сокращает продолжительность каждого цикла ПЦР.
Современные термоциклеры приспособлены для использования специальных тонкостенных пластиковых пробирок для реакционной смеси, что позволяет ускорить теплообмен между блоком прибора и реакционной смесью и в конечном итоге дополнительно сократить время проведения реакции.
Таким образом, стандартная ПЦР может быть осуществлена за 1 – 3 ч. Многие приборы позволяют программировать специальные усложненные температурные профили, необходимые для специфических модификаций процесса ПЦР.
Другие модификации ПЦР:
- Мультиплексная ПЦР
- Ассиметричная ПЦР
- ПЦР с обратной транскрипцией (ОТ-ПЦР) (амплификация РНК)
- Амплификация протяжённых участков ДНК (до 40 т.п.н.)
- Полногеномная амплификация
- RAPD ПЦР
- Количественная ПЦР в реальном времени
Параллельно с усовершенствованием технологии ПЦР развивались и методы анализа продуктов реакции. Метод гель-электрофореза с последующим окрашиванием красителем, специфичным к ДНК, например бромистым этидием, традиционно применяется во многих лабораториях для обнаружения амплифицированной ДНК и определения ее размера.
Использование гибридизации с внутренними ДНК-зондами позволяет в ряде случаев значительно повысить чувствительность и специфичность детектирования ПЦР-продуктов. Благодаря отсутствию необходимости в подготовке и проведении электрофоретического разделения, возможности автоматизации для анализа большого количества образцов и использования нерадиоактивного формата детектирования, этот метод становится все более распространенным. В некоторых случаях применение специальных флюоресцентных «маркеров» позволяет контролировать проведение амплификации или детектирование конечных продуктов ПЦР непосредственно в реакционной пробирке.
Увидеть результаты ПЦР очень просто: достаточно провести электрофорез реакционной смеси после ПЦР, и будет видна яркая полоса с полученными копиями. Метод гель-электрофореза с последующим окрашиванием красителем, специфичным к ДНК, традиционно применяется во многих лабораториях для обнаружения амплифицированной ДНК и определения ее размера.
Можно еще много говоритьо различных тонкостях ПЦР, но важнее всего сказать об альтернативных классическому форезу методах определения результатов. Например, довольно очевидным вариантом является добавление в реакционную пробирку перед началом реакции веществ, флуоресцирующих в присутствии ДНК. Тогда, сравнив изначальную флуоресценцию с конечной, можно увидеть, синтезировалось ли значительное количество ДНК или нет. Но этот способ не специфичен: мы никак не сможем определить, синтезировался ли необходимый фрагмент, или это какие-то праймеры слиплись и достроились до непредсказуемых последовательностей.
ПЦР в реальном времени и количественная ПЦР
quantitative and real-time PCR
 Наиболее интересным вариантом является ПЦР «в реальном времени» («real-time PCR»). Существует несколько реализаций этого метода, но идея везде одна и та же: можно прямо в ходе реакции наблюдать за накоплением продуктов ПЦР (по флуоресценции). Соответственно, для проведения ПЦР «в реальном времени» нужен специальный прибор, способный возбуждать и считывать флуоресценцию в каждой пробирке. Самое простое решение — добавить в пробирку те же самые вещества, которые флуоресцируют в присутствии ДНК, однако минусы такого метода уже были описаны выше.
Наиболее интересным вариантом является ПЦР «в реальном времени» («real-time PCR»). Существует несколько реализаций этого метода, но идея везде одна и та же: можно прямо в ходе реакции наблюдать за накоплением продуктов ПЦР (по флуоресценции). Соответственно, для проведения ПЦР «в реальном времени» нужен специальный прибор, способный возбуждать и считывать флуоресценцию в каждой пробирке. Самое простое решение — добавить в пробирку те же самые вещества, которые флуоресцируют в присутствии ДНК, однако минусы такого метода уже были описаны выше.
Строго это называется «ПЦР с регистрацией флуоресценции в режиме реального времени». - Ред.
Рисунок 30 (слева). Схема работы ПЦР «в реальном времени»: Taq Man Assay. (слева).
Самой популярной реализацией такого подхода является метод выщепления флуорофора за счет разрушения зонда (TaqMan Assay; рис.30). В этом случае в реакционной смеси должен присутствовать еще один компонент - специальный одноцепочечный ДНК-зонд: молекула ДНК, комплементарная последовательности амплифицируемого фрагмента, расположенной между праймерами. При этом к одному его концу должен быть химически приделан флуорофор (флуоресцирующая молекула), а к другому – гаситель (молекула, поглощающая энергию флуорофора и «гасящая» флуоресценцию). Когда такой зонд находится в растворе или комплементарно связан с целевой последовательностью, флуорофор и гаситель находятся относительно недалеко друг от друга, и флуоресценции не наблюдается. Однако за счет 3´-экзонуклеазной активности, которой обладает Taq-полимераза (то есть она расщепляет ДНК, на которую «натыкается» в ходе синтеза, и на ее месте синтезирует новую), зонд при синтезе второй цепи разрушается, флуорофор и гаситель за счет диффузии удаляются друг от друга, и появляется флуоресценция.

Рисунок 31. Пример кривых накопления флуоресценции в ПЦР «в реальном времени» (в количественной ПЦР в реальном времени): зависимость интенсивности флуоресценции (в нескольких пробирках — на каждую своя кривая) от номера цикла.
Поскольку число копий в ходе ПЦР растет экспоненциально, так же растет и флуоресценция. Однако это продолжается недолго, поскольку в какой-то момент эффективность реакции начинает падать из-за постепенной инактивации полимеразы, нехватки каких-то компонентов и т.п. (рис. 31).
Анализируя графики роста флуоресценции, можно много понять о протекании ПЦР, но, самое важное, можно узнать, сколько ДНК-матриц было изначально: это так называемая количественная ПЦР (quantitative PCR, qPCR). Количественная ПЦР (греч. polymeres — состоящий из многих частей, многообразный; лат. re- — приставка, обозначающая повторность действия, и actio — действие) - вариант ПЦР, при котором непрерывно регистрируется кинетика реакции, что позволяет количественно оценивать содержание определенных нуклеотидных последовательностей ДНК в сложной смеси молекул.
6. Секвенирование ДНК по Сэнгеру
Рис. 32: биофизик Фредерик Сэнгер - создатель популярного метода секвенирования биополимеров, считающегося золотым стандартом в данной сфере.
1. Метод «плюс-минус»
Можно сказать, важнейшие методы манипуляции с ДНК уже описаны. Следующий этап - определение собственно нуклеотидной последовательности цепи в молекуле - секвенирование. Секвенирование ДНК относится к методам определения порядка расположения нуклеотидных оснований аденина, гуанина, цитозина и тимина в молекуле ДНК.
Один из наиболее популярных методов секвенирования обязан своим появлением английскому биофизику Фредерику Сэнгеру (1918–2013) - единственному ученому в истории мировой науки, получившему сразу две Нобелевские премии по химии (в 1958 и 1980 годах). Первую премию присудили за установление структур белков, особенно инсулина, а вторую награду ему вручили в том числе и за разработку методов определения первичной последовательности нуклеиновых кислот.
Методику секвенирования ДНК с использованием радиоактивно меченых нуклеотидов и ДНК-полимеразы предложили Сэнгер и его коллеги в 1977 году, причем с течением времени этот метод прошел несколько модификаций и к настоящему моменту считается золотым стандартом современного секвенирования.

Рисунок 33. «Плюс-минус» метод секвенирования ДНК
Первоначально Ф. Сэнгер и Алан Коулсон разработали так называемый «плюс-минус» метод секвенирования ДНК, который можно подразделить на две основные стадии:
1. Полимеразная цепная реакция, в которой используется ДНК, фермент (ДНК-полимераза), олигонуклеотидные праймеры и смесь четырех дезоксинуклеотидов (dNTPs) (А, Т, G и C), причем один из дезоксинуклеотидов радиоактивно помечен по α-положению фосфата (32P).
2. Очистка смеси амплифицированных фрагментов от дезоксинуклеозидтрифосфатов, не вступивших в реакцию (например, на колонках). Смесь делят на восемь равных частей (в разных пробирках). В «плюс»-системе проводят четыре ПЦР-реакции в присутствии каждого из четырех типов дезоксинуклеозидтрифосфатов; параллельно в «минус»-системе проводят четыре ПЦР-реакции в отсутствии каждого из них. Далее результаты визуализируют с помощью электрофореза, и определяют последовательность ДНК, исходя из того, что в «плюс»-системе терминация (прерывание) ПЦР происходит после конкретного dNTP, а в «минус»-системе — перед ним.
2. Метод «терминаторов»

Рис. 34. Метод терминирующих аналогов трифосфатов. Ферментативный метод секвенирования Сэнгера с дидеокси- обрывом цепи основан на линейной амплификации одноцепочечной матричной ДНК с использованием 5'-флуоресцентно меченого праймера и модифицированной Taq полимеразы. Синтез комплементарной ДНК-цепи начинается на специфическом праймер-сайте и заканчивается включением обрывающего цепочку ddNTP, который случайным образом введен вместо соответсвующего dNTP. При использовании четырех разных ddNTP в четырех отдельных реакционных пробирках, получается набор цепей удлиненных праймеров, терминированных в каждой позиции A, C, G и T. Когда эти фрагменты разделены на подходящей гелевой матрице, последовательность ДНК определяется по порядку смещения полос (снизу вверх).
 В 1977 г. автор «плюс-минус» метода предложил еще один способ ферментативного секвенирования, получивший название метода терминирующих аналогов трифосфатов (синонимы метода «терминаторов» и метод «обрыва цепи»). Более мощный и более технологичный, этот способ, несколько модифицированный, применяется до сих пор. В основе метода так же лежало ферментативное копирование с помощью фрагмента ДНК полимеразы. В качестве праймеров использовали синтетические олигонуклеотиды. Специфическую терминацию (прекращение) синтеза обеспечивали добавлением в реакционную смесь помимо четырех типов dNTPs (А, Т, G и C) (один из которых был радиоактивно мечен по альфа положению фосфата) еще и одного из 2',3'-дидезокси-нуклеозидтрифосфатов (ddATP, ddTTP, ddCTP или ddGTP), который способен включаться в растущую цепь ДНК, но не способен обеспечивать дальнейшее копирование из-за отсутствия 3'-ОН группы. Отношение концентраций dNTP/ddNTP авторы подбирали экспериментально, так, чтобы в итоге получить набор копий ДНК различной длины.
В 1977 г. автор «плюс-минус» метода предложил еще один способ ферментативного секвенирования, получивший название метода терминирующих аналогов трифосфатов (синонимы метода «терминаторов» и метод «обрыва цепи»). Более мощный и более технологичный, этот способ, несколько модифицированный, применяется до сих пор. В основе метода так же лежало ферментативное копирование с помощью фрагмента ДНК полимеразы. В качестве праймеров использовали синтетические олигонуклеотиды. Специфическую терминацию (прекращение) синтеза обеспечивали добавлением в реакционную смесь помимо четырех типов dNTPs (А, Т, G и C) (один из которых был радиоактивно мечен по альфа положению фосфата) еще и одного из 2',3'-дидезокси-нуклеозидтрифосфатов (ddATP, ddTTP, ddCTP или ddGTP), который способен включаться в растущую цепь ДНК, но не способен обеспечивать дальнейшее копирование из-за отсутствия 3'-ОН группы. Отношение концентраций dNTP/ddNTP авторы подбирали экспериментально, так, чтобы в итоге получить набор копий ДНК различной длины.
Таким образом, для определения первичной структуры исследуемого фрагмента ДНК требовалось провести четыре реакции копирования: по одному типу терминаторов в каждой из реакций (т.е. в каждой из 4 пробирок свой терминатор). После этого полученные продукты разгонялись в полиакриламидном геле на соседних дорожках и по расположению полос определялась последовательность нуклеотидов.

Рисунок 35. Метод «терминаторов». В каждой пробирке образуется набор фрагментов ДНК разной длины, которые заканчиваются одним и тем же нуклеотидом. Затем полученные фрагменты визуализируют с помощью электрофореза и, сравнивая длины фрагментов из четырех реакций с ddATP, ddTTP, ddCTP или ddGTP, восстанавливают последовательность ДНК
Выполнение метода «терминаторов»:
- Ключевым принципом метода Сенгера было использование дидезоксинуклеозидтрифосфатов (ddNTPs) или сокр. дидезоксинуклеотидов в качестве Терминаторов цепей ДНК.
- Метод прерывания цепи требует одноцепочечного шаблона ДНК, ДНК-праймера, ДНК-полимеразы, радиоактивно или флуоресцентно меченых нуклеотидов и модифицированных нуклеотидов, которые прекращают удлинение нити ДНК.
- Образец ДНК разделен на четыре отдельные реакции секвенирования, содержащие все четыре стандартных дезоксинуклеотида (dATP, dGTP, dCTP, dTTP) и ДНК-полимеразу.
- К каждой реакции добавляется только один из четырех ddNTPs (ddATP, ddGTP, ddCTP, ddTTP), которые представляют собой цепь, заканчивающуюся нуклеотидами, в которой отсутствует группа 3’-OH, необходимая для образования фосфодиэфирной связи между двумя нуклеотидами, что приводит к прекращению удлинения нити ДНК и получению фрагментов ДНК различной длины.
- Вновь синтезированные и меченые фрагменты ДНК денатурируются нагреванием и разделяются по размеру методом гель-электрофореза на денатурирующем полиакриламидном геле с каждой из четырех реакций, протекающих в одной из четырех отдельных полос (полосы A, T, G, C).
- Полосы ДНК затем визуализируются с помощью авторадиографии или ультрафиолетового излучения, и последовательность ДНК может быть непосредственно считана с рентгеновской пленки или изображения геля.
- Темная полоса на полосе указывает на фрагмент ДНК, который является результатом разрыва цепи после включения ddNTPs (ddATP, ddGTP, ddCTP или ddTTP).
- Относительное положение различных полос между четырьмя полосами используется затем для считывания (снизу вверх) последовательности ДНК.
- Технические варианты секвенирования конца цепи включают маркировку нуклеотидами, содержащими радиоактивный фосфор для маркировки, или использование праймера, меченного на 5’ конце флуоресцентным красителем.
- Секвенирование красителя-праймера облегчает считывание в оптической системе для более быстрого и экономичного анализа и автоматизации.

Ключевая характеристика метода прерывания цепи:
- Использует дидезоксинуклеозидтрифосфаты (2’,3’-dideoxynuceloside triphosphate или ddNTPs) для прекращения синтеза ДНК.
- Реакции синтеза ДНК в четырех отдельных пробирках
- Радиоактивный dATP также входит во все трубки, поэтому продукты ДНК будут радиоактивными.
- Получается ряд фрагментов ДНК, размеры которых можно измерить методом электрофореза.
- Последняя база в каждом из этих фрагментов известна.
Методы прерывания цепи упростили секвенирование ДНК.
Ограничения:
- Неспецифическое связывание праймера с ДНК, влияющее на точное считывание последовательности ДНК.
- Вторичные структуры ДНК влияют на верность последовательности.
Автоматизированные модификации метода «терминаторов» активно применяют до сих пор в специальных приборах - секвенаторах. Открытие многочисленных флуоресцентных молекул позволило отказаться от использования радиоактивной метки и сделало возможным проведение реакции в одной пробирке. Реакционную смесь разделяют капиллярным электрофорезом, a выстроившиеся в синтезируемую цепочку ДНК меченые нуклеотиды затем регистрируют детекторами флуоресценции, предоставляя возможность считывать последовательность всего секвенируемого ДНК-фрагмента.
Использование секвенирования по Сэнгеру
Секвенирование по Сэнгеру позволяет «считывать» последовательности до 1000 пар нуклеотидов и применяется для небольших фрагментов генома/генов. В частности, оно используется для:
- секвенирования отдельных участков генома с целью анализа мутаций и полиморфизмов;
- идентификации вирусов и организмов (бактерий, растений, грибов и животных);
- валидации данных, полученных на платформах секвенирования нового поколения (NGS);
- микросателлитного анализа;
- анализа делеций и инсерций (малых и протяженных).
Наиболее популярными секвенаторами, использующими технологию секвенирования по Сэнгеру, являются приборы, производимые компанией Thermo Fisher Scientific: 3730xL, 3730, 3500xL, 3500, 3130xL, 3130, 310.
Следует отметить, что все описанные выше типы исследований сейчас можно проводить с помощью секвенирования «нового поколения», однако главные плюсы секвенирования по Сэнгеру - высокая точность (достоверность) полученных данных и невысокая стоимость работ при анализе небольшого количества ДНК-фрагментов - сохраняют актуальность этого типа определения последовательности НК.
7. СЕКВЕНИРОВАНИЕ НОВОГО ПОКОЛЕНИЯ

NGS: ВЫСОКОПРОИЗВОДИТЕЛЬНОЕ СЕКВЕНИРОВАНИЕ
New generation sequencing, NGS
Как работает NGS. NGS – это группа методов основанных на одновременном секвенировании (параллельном прочтении) множества индивидуальных молекул ДНК (от деятков миллионов до миллиардов). Вся технология NGS упрощенно состоит из трех основных этапов, которые могут отличаться в той или иной мере друг от друга в зависиморсти о вида используемой платформы севенирования:
I. Подготовка библиотек (матриц), оценка их качества
а) Выделение ДНК или РНК
б) Разрушение ДНК на короткие участки (УФ-излучением, ферментами)
в) Достройка ДНК под требования платформы секвенирования
г) Предамплификация – формирование молекулярных кластеров
II. Секвенирование (виды)
а) Сенквенирование путем синтеза - Одновременное секвенирование миллионов коротких фрагментов ДНК через синтез – получение миллионов одиночных чтений (ридов). Процесс предусматривает использование РНК-зависимой ДНК-полимеразы и праймеров для создания новой комплементарной цепи ДНК (циклический процесс включения нуклеотидов в цепь детектируется).
б) Секвенирование лигированием - Процесс секвенирования ДНК с использованием ДНК-лигазы для создания новой комплементарной нити ДНК.
в) Секвенирование с помощью нанопор - Принцип детектирования основан на контроле ионного тока, проходящего через нанопору при подаче напряжения на мембрану.
III. Анализ данных – биоинформатика
а) Фильтрация ридов, построение метрик качества
б) Сборка – выравнивание ридов так, чтобы они сформировали разумную последовательность
в) Анализ биологического смысла данных

Примечание редактора
Как было уже отмечено выше, если имеется желание ознакомиться с темой секвенирования (в данном случае с NGS) более детально, а не в обзорном порядке, то в данном разделе предусмотрен т.н. "материал в помощь", где в трех литературных источниках и в видеолекциях можно найти всю интересующую информацию к темам этой страницы.
Некоторые технологии (платформы) секвенирования NGS
Платформа Illumina

Рис. 37. Оптическая детекция результатов анализа на платформе Illumina
При помощи сэнгеровского секвенирования расшифрован геном человека, но сейчас применяются секвенаторы нового поколения. В них участки ДНК многократно клонируются, но процесс чтения происходит по-другому. Один из новых методов - это секвенирование по методу Solexa (ныне Illumina).
ДНК фрагментируется. К каждому участку ДНК с двух сторон добавляют адаптеры - известные небольшие последовательности нуклеотидов. Затем смесь помещается на подложку, на которой в виде решётки размещены участки ДНК, комплементарные адаптерам. Кроме того, адаптеры содержат праймеры для ДНК-полимеразы, которая осуществляет репликацию ДНК. На подложке фрагменты ДНК многократно клонируют, получая кластеры. ДНК привязана к подложке сразу двумя концами. ДНК денатурируют. Подложка помещается в раствор, содержащий ДНК-полимеразу и помеченные нуклеотиды. После присоединения комплементарного нуклеотида лишние нуклеотиды смывают, а метки присоединившихся считывают. В технологии Illumina это флуоресцентные метки, которые можно заставить светиться разным цветом и сфотографировать. Затем ДНК обрабатывается фосфатазой и процесс повторяется. В результате на каждом цикле мы прочитываем одновременно очень большое число нуклеотидов из разных последовательностей. После секвенирования множества коротких фрагментов они собираются в единую молекулу ДНК.



Рис. 38. Схема Illumina-секвенирования. Первый этап этого метода, заключается в формировании библиотеки фраментов ДНК с «привязанными» на обоих концах адапторами. На этапе амплификации технология применяет поверхность ячеек, на которые нанесены в смеси те же адаптеры; молекулы ДНК в ходе ПЦР образуют «мостики», поэтому такой способ носит название «мостиковой амплификации» (bridge amplification). Секвенирование образующихся кластеров в ДНК проходит в ячейках с участием обратимо меченых нуклеотидов (reversible dye terminator), которые добавляют последовательно в циклическом режиме. Т.е. каждое событие включения меченого нуклеотида регистрирует оптическое устройство, после чего нуклеотид деблокируют, и синтез продолжается.
Итак, этапы секвенирования Illumina такие:
1. Сначала создается библиотека фрагментов ДНК из секвенируемого генома (или любого другого источника ДНК). ДНК с помощью ультразвука или специального фермента расщепляется на произвольные фрагменты длиной в несколько сотен нуклеотидов, из которых выбираются обладающие заданной длиной (выбирается экспериментатором). После этого к ним с двух концов ковалентно присоединяются различные адаптерные последовательности;
2. Этот раствор поступает на специальный микрочип с «пришитыми» к нему фрагментами ДНК, комплементарными адаптерным последовательностям, - праймерами. Там участки геномной ДНК прикрепляются с помощью адаптерных последовательностей к молекулам ДНК на чипе. С помощью ДНК-полимеразы у всех таких фрагментов достраивается вторая комплементарная цепь, которая будет уже ковалентно связана с праймером. Она случайно изгибается, и в какой-то момент попадет на праймер, комплементарный ее адаптерной последовательности. Они свяжутся, и снова достроится вторая цепь. Это повторяют несколько раз, после чего один из типов праймеров отрезают от молекулы ДНК, чтобы в образовавшемся кластере остались только одинаковые цепи. Всего на чипе образуются сотни миллионов таких кластеров;
3. Собственно секвенирование. К кластерам добавляют праймеры, комплементарные одной из адаптерных последовательностей, с которой они связываются. Затем добавляют ДНК-полимеразу и специально модифицированные нуклеотиды с прикрепленным к ним флуорофором (разным у разных типов нуклеотидов), синтез после которых заблокирован. Они присоединяются к молекулам ДНК в соответствии с правилом комплементарности. Затем камера сканирует, какой флуорофор появился в каждом кластере по цвету свечения в лазере, и компьютер запоминает расположение кластеров. По этому определяется, какой нуклеотид встроился в цепь. После этого отщепляются флуорофоры от всех нуклеотидов, и дальнейший синтез цепи разблокируется. Все повторяется снова: добавляются нуклеотиды, чип сканируется, чтобы определить, какой нуклеотид присоединился к какому кластеру и т.п.. Таким образом секвенируют до 100 нуклеотидов в каждом кластере, а всего их сотни миллионов — в итоге это десятки миллиардов пар нуклеотидов.
Видео: Процесс секвенирования Illumina
Технологии SLR на платформах Illumina / 10XGenomics
Технологии SLR (synthetic long read - синтезируемые длинные последовательности) В отличие от истинных платформ секвенирования с длительным считыванием, основана на системе штрих-кодирования для связывания фрагментов, которые секвенируются на существующих секвенсорах для чтения коротких последовательностей. В настоящее время существуют две системы: платформа секвенирования Illumina SLR (ранее Moleculo; рисунок 39 A) и система на основе эмульсии 10X Genomics (рисунок 39 B).
В системе Illumina геномная ДНК разрезается на фрагменты по 8–10 кб (килобаз) после чего происходит лигирование с образованием «длинного фрагмента». Эти большие фрагменты затем разделяют на микротитрационный планшет (3000 фрагментов в 1 лунке) и подвергают дальнейшей фрагментации и добавлению адаптера посредством тагментации. Каждая лунка содержит один штрих-код. Последовательности ДНК объединяют в пул и проводят секвенирование с последующей локальной сборкой для восстановления исходных длинных фрагментов. С помощью секвенирования на основе эмульсии 10X Genomics природные фрагменты ДНК размером до 100 кб смешиваются в мицеллах в эмульсии вместе с гелевыми шариками, содержащими последовательности адаптера и штрих-кода (рисунок 39B). Благодаря микроструйной технологии 10X Genomics требуется очень небольшое количество исходного материала (1 нг). В каждой мицелле шарик геля растворяется, и более мелкие фрагменты ДНК амплифицируются из исходных больших фрагментов, каждый со штрих-кодом, идентифицирующим исходную мицеллу. Затем фрагменты со штрих-кодом объединяют, а затем проводят классическую подготовку и секвенирование библиотеки Illumina. Полученные последовательности собирают, чтобы сформировать серию стабильных фрагментов по 50 кб.
Рисунок 39. Схематичное описание синтетических технологий секвенирования длинных последовательностей
(A) Рабочий процесс синтетической технологии Long-read от lllumina. Геномная ДНК разрезается на 10-килобайтные фрагменты с последующим лигированием с «длинными фрагментами» адаптеров. Полученные фрагменты распределяют в 384-луночном планшете (3000 молекул на лунку) с последующей ПЦР длинных фрагментов. В последующем процессе «тагментирования» (технология Nextera от Illumina) продукты ПЦР дополнительно фрагментируются, и одновременно добавляются последовательности адаптера. Затем меченую ДНК снова амплифицируют с помощью ПЦР и уникального индекса, и последовательности адаптера P5 и P7 добавляют в каждую лунку 384-луночного планшета. Эти конечные продукты объединяются и секвенируются по технологи Illumina.
(B) 10X Genomics — эмульсионное секвенирование. Прибор Chromium может разделять природные фрагменты ДНК на 1 миллион мицелл («GEM») вместе со штрих-кодированными праймерами (доступно 4 миллиона штрих-кодов) и реагентами для ПЦР. В каждой мицелле меньшие фрагменты ДНК амплифицируются из исходного большего фрагмента, каждый со штрих-кодом, идентифицирующим исходную мицеллу. Получающиеся меньшие фрагменты объединяются и секвенируются по механизму Illumina. После секвенирования последовательности упорядочены и связаны друг с другом, образуя серию стабильных фрагментов в диапазоне 50 кб.
Быстрота и дешевизна методов секвенирования нового поколения, недоступная ранее, спровоцировала бум в индустрии геномных исследований. Появилась возможность проводить ранее технически недоступные эксперименты.
Пиросеквенирование
Пиросеквенирование – определение последовательности нуклеотидов в ДНК по принципу «секвенирование путем синтеза». При включении нуклеотида во вновь синтезируемую последовательность ДНК высвобождается пирофосфат, который регистрируется посредством хемилюминесценции. Поскольку ДНК иммобилизована, растворы нуклеотидов A, C, G и T добавляют и отмывают поэтапно, после каждой реакции.

Последовательность растворов, которые дают хемилюминесцентный сигнал, позволяет определить последовательность ДНК. Когда полимераза включает комплементарный нуклеотид и высвобождается пирофосфат (PPi), фермент АТФ-сульфурилаза превращает PPi в аденозинтрифосфат (АТФ или англ. ATP) в присутствии аденозин-5´-фосфосульфата. В присутствии АТФ люцифераза превращает люциферин в оксилюциферин с высвобождением видимого света, который регистрируется и анализируется компьютерной программой. Ораничением в применении этой технологии является длина последовательности нуклеотидов, которая составляет около 300-500 нуклеотидов.
Технология пиросеквенирования позволяет получать информацию о 400 миллионах нуклеотидах за 10-часовой период работы на одной установке. Методология включает следующие этапы (Рис. 41).

Рис. 41. Схема пиросеквенирования
А - ДНК фрагментируется, к фрагментам пришиваются олигонуклеотиды-«адаптеры»; полученные двуцепочечные молекулы ДНК разделяются на две комплементарные цепи. Б - Одноцепочечные молекулы ДНК прикрепляются к бусинкам в условиях, стимулирующих попадание лишь одной молекулы на бусинку. Отдельные бусинки заключаются в капли реакционной смеси, окруженные маслом. Количество молекул на бусинке увеличивается в миллионы раз в результате эмульсионной полимеразной цепной реакции (эПЦР). В - Эмульсия разбивается, и цепи ДНК-фрагментов, образовавшиеся в результате эПЦР, разделяются. Бусинки, несущие на своей поверхности миллионы одноцепочечных копий первоначального фрагмента ДНК, помещаются в лунки оптико-волоконного слайда, по одной в каждую лунку. Г - В каждую лунку добавляются бусинки поменьше, несущие на своей поверхности ферменты, необходимые для пиросеквенирования. Д - Микрофотография эмульсии, изображающая «пустые» капли и капли, содержащие бусинки с ДНК-матрицей. Толстая стрелка указывает на 100-мкм каплю, тонкая - на 28-мкм бусинку. Е - Микрофотография фрагмента оптико-волоконного слайда, полученная при помощи сканирующего электронного микроскопа. Видна плакировка оптических волокон и пустые лунки.
ДНК, фрагментируют на кусочки по 300–500 пар оснований. Затем комплементарные цепи денатурируют, к каждой цепи фрагментов ДНК пришивают одинаковый для всех олигонуклеотид-«адаптер», который позволяет прикрепиться на пластиковые бусинки. Смесь фрагментов ДНК разбавляют так, что каждая бусинка получает по одной индивидуальной цепи. Затем каждая бусинка оказывается заключённой в капельку, окруженную маслом и содержащую смесь для полимеразной цепной реакции, которая проходит в каждой капельке эмульсии (эмульсионная ПЦР). Это приводит к «клональной амплификации» цепей ДНК, на поверхности бусинки удерживается уже не одна, а около 10 млн копий («клонов») уникальной ДНК-матрицы.

Рис. 42. Дополнительная визуализация к рисунку 41.
Далее двуцепочечные фрагменты ДНК, образовавшиеся в ходе ПЦР, разделяются и бусинки, несущие одноцепочечные копии ДНК-матрицы, помещают в лунки. Каждая лунка - это отдельный пиколитровый «реактор», в котором идет реакция секвенирования. Происходит поочередное добавление и удаление реагентов и продуктов реакции. Помимо бусинок с ДНК-матрицей, в каждую лунку добавляют более мелкие бусинки с иммобилизованными на поверхности ферментами, необходимыми для пирофосфатного секвенирования. Нуклеотиды подаются последовательно в проточную камеру, куда помещается слайд с лунками. Когда определённый нуклеотид встраивается в растущую цепь ДНК в лунке высвобождается молекула пирофосфата, которая является предшественником компонента другой ферментативной реакции. Её катализирует фермент - люцифераза светлячка Photinus pyralis. Для её осуществления необходим аденозинтрифосфат (АТФ). Пирофосфат превращается в АТФ под действием фермента АТФ-сульфурилазы. В присутствии АТФ фермент люцифераза окисляет люциферин до оксилюциферина, эта реакция сопровождается хемилюминесценцией (вспышкой света), которая регистрируется для каждой индивидуальной лунки, в которой произошло встраивание известного нуклеотида.
Платформа 454 Roche
Самый известный из примеров пиросеквенирования - это платформа 454 Roche, основанная на ультраглубоком пиросеквенировании (ultra-deep pyrosequencing).
Анализ включает в себя следующие стадии:
I. Приготовление библиотеки фрагментов (shot-gun);
- двуцепочечную ДНК разрушают до фрагментов размером 300-500 н.п.
- присоединение меченых биотином праймеров и адаптеров; к обоим концам фрагментов «пришивают» короткие олигонуклеотиды, необходимые для обеспечения комплементарных взаимодействий на последующих этапах;
- двуцепочечную ДНК «расщепляют» на одноцепочечные молекулы.
 II. Эмульсионная ПЦР: фрагменты ДНК вместе с реагентами ПЦР и специальными шариками, покрытыми стрептавидином для захвата ДНК (beads), смешивают в водном растворе и вносят в пробирки с синтетическим маслом. После интенсивного перемешивания формируется эмульсия «вода в масле», состоящая из крошечных капелек. Концентрация шариков и ДНК подбирается таким образом, чтобы в каждую из этих капелек попала и адсорбировалась на шарике только одна молекула ДНК-фрагмента. В ходе реакции ПЦР каждая капелька выступает в роли «реактора», продуцирующего только один вид ампликонов в количестве десятков миллионов копий.
II. Эмульсионная ПЦР: фрагменты ДНК вместе с реагентами ПЦР и специальными шариками, покрытыми стрептавидином для захвата ДНК (beads), смешивают в водном растворе и вносят в пробирки с синтетическим маслом. После интенсивного перемешивания формируется эмульсия «вода в масле», состоящая из крошечных капелек. Концентрация шариков и ДНК подбирается таким образом, чтобы в каждую из этих капелек попала и адсорбировалась на шарике только одна молекула ДНК-фрагмента. В ходе реакции ПЦР каждая капелька выступает в роли «реактора», продуцирующего только один вид ампликонов в количестве десятков миллионов копий.
III. Собственно секвенирование проводят в планшете, содержащем до полутора миллионов микрообъемных ячеек.
В каждую ячейку помещается только один шарик; кроме него, там же находятся ДНК-полимераза и еще два фермента - сульфурилаза и люцифераза, а также субстрат, которые генерируют люминесцентный сигнал на выходе реакции.
Сама реакция секвенирования основана на последовательном включении нуклеотидов в растущую цепочку ДНК, при этом растворы нуклеотидов пропускают через ячейки планшета по очереди, каждый раз отмывая несвязанные молекулы.
Если при очередном пропускании в ячейке происходит присоединение нуклеотида, вырабатывается световой сигнал, если не происходит - сигнал отсутствует. Каждый «нуклеотидный раунд» сопровождается регистрацией сигнала, причем оптическая часть системы устроена таким образом, что сигнал от каждой ячейки регистрируется и записывается отдельно. Интенсивность сигнала пропорциональна числу включенных нуклеотидов в одном раунде (например, если последовательность содержит три G подряд, то при пропускании G будет получен сигнал, втрое превышающий сигнал от включения одного G).
IV. Результат анализа последовательности в индивидуальной ячейке называется флоуграммой (flowgram) и выглядит как последовательность столбиков, цвет которых соответствует одному из нуклеотидов Т, С, А или G; например, на рисунке изображена флуограмма последовательности, которую следует читать как TCAGATCCGTGGGTG.
V. Последним этапом секвенирования является совмещение данных, полученных при анализе индивидуальных фрагментов. Для этого в каждом случае применяется специально разработанное для каждой платформы программное обеспечение, которое объединяет данные от каждой молекулы ДНК в единую последовательность (общее для всех - ред.)
Технические характеристики имеющихся сегодня платформ для секвенирования NGS отличаются, так же как и длина прочтений (reads); наибольшая длина у Roche (400-500 нуклеотидов); lllumina и SOLiD (см. ниже) пока прочитывают от 35 до 50 нуклеотидов, поэтому при решении конкретных задач приходится учитывать эти, а также многие другие параметры. Кроме этих трех методов, имеется в распоряжении исследователей и разрабатывается множество других, основанных на оригинальных подходах, например, lonTorrent (Life Technologies), Helicos (BioSciences Corporation), MinlON (Oxford Nanopore Technologies) и т.д.
ОСНОВНЫЕ ТЕХНОЛОГИИ NGS
I. Секвенирование синтезом
Платформы для технологии секвенирования синтезом
|
Создатель
(производитель)
|
Амплификация
(увеличение числа копий участков ДНК)
|
Детекция
(система обнаружения)
|
|
Секвенирование по Сэнгеру
|
Одномолекулярная
|
Оптическая
|
|
Roche 454 (GS FLX)
|
Клональная
|
Оптическая
|
|
Helicos BioSciences (Heliscope)
|
Одномолекулярная
|
Оптический
|
|
Ion Torrent (ThermoFisher)
|
Клональная
|
Полупроводниковая
|
|
Illumina
|
Клональная
|
Оптическая
|
|
PacBio
|
Одномолекулярная
|
Оптическая
|
|
GeneReader (Qiagen)
|
Клональная
|
Оптическая
|
|
GenapSys (GENIUS)
|
Клональная
|
Полупроводниковая
|
|
||
Одномолекулярное секвенирование Helicos BioSiences - метод секвенирования единичных молекул, разработанный HeliScope (Helicos BioSciences) даёт информации около 1Gb/день. Принцип работы: после клональной амплификации образца происходит фрагментация ДНК с последующим полиаденилированием на 3'-конце с дальнейшим секвенированием, чередующимся с промыванием образцов нуклеотидами с флуоресцентной меткой.
Одномолекулярное секвенирование в реальном времени Pacific BioSciences (Single molecule real time sequencing, SMRT) основано на наблюдении за работой единичной молекулы ДНК-полимеразы. Использование четырех флюоресцентно меченных нуклеотидов позволяет определить какой нуклеотид присоединяет ДНК-полимераза в данный момент времени. Т.е. как только ДНК-полимераза добавит в последовательность правильный нуклеотид, он излучает свет с определенной длиной волны. Детектор внутри волновода (ZMW, камера нанофотонической визуализации) захватывает испущенный свет.
Метод секвенирования Ion Torrent Sequencing - основан на связи между химической и цифровой информацией, что позволяет быстро секвенировать большое количество образцов. Технология также называется рН-индуцированным секвенированием. Процесс основан на детекции протонов, которые получаются при синтезе цепи ДНК как побочный продукт. Как следствие, рН раствора меняется, что и детектируют.
Introduction to Ion Torrent™ Next-Generation Sequencing Technology
Видео: Технология Ion Torrent™ непосредственно преобразует химически закодированную информацию (A, C, G, T) в цифровую информацию (0, 1) на полупроводниковом чипе. Этот подход соединяет простую химию с запатентованной полупроводниковой технологией. Результатом является технология секвенирования, которая является более простой, быстрой, экономически эффективной и масштабируемой, чем многие другие технологии секвенирования следующего поколения. (Если виджео не отображается см. по ссылке →)
- Каждый нуклеотид заполняет чип последовательно
- Когда нуклеотид добавляется к матрице ДНК и затем включается в цепочку ДНК, высвобождается ион водорода (H+).
- Заряд от этого Иона изменяет рН раствора, который может быть обнаружен датчиком ионов.
- Секвенсор вызывает базу, переходя непосредственно от химической информации к цифровой информации.
- Если следующий нуклеотид, который заполняет чип, не совпадает, не будет записано изменение напряжения и не будет вызвана база.
- Если на нити ДНК есть два одинаковых основания, напряжение будет удвоено, и чип запишет два одинаковых основания.
Платформа Ion Torrent отличается от остальных технологий секвенирования тем, что в ней не используются модифицированные нуклеотиды и оптические методы. Метод Ion Torrent позволяет исследовать транскриптомы, малые РНК, проводить ChIP-seq. Более того, с его помощью можно изучать геномы микробных сообществ.
Оптическая детекция

1. Четырехканальный метод SBS
- При 4-канальном SBS (секвенировании синтезом) каждый цикл секвенирования требует четырех флуоресцентных красителей для каждого основания и четырех изображений для определения последовательности ДНК.
- Системы MiSeqTM и HiSeqTM используют четырехканальную химическую технологию
2. Примерно в 2014 году Illumina перешла с 4-канального метода SBS на 2 - канальный метод SBS. Здесь теперь тимины помечены зеленым флуорофором, цитозины - красным флуорофором, а аденины – как красными, так и зелеными флуорофорами. Гуанины постоянно темные.
- Изображения принимаются с использованием красных и зеленых полос фильтра (2 изображения за цикл, вместо 4).
- Это ускоряет секвенирование и время обработки данных, обеспечивая при этом одинаковое качество и точность.
- Системы MiniSeqTM, NextSeqTM и NovaSeqTM используют двухканальную технологию.
3. В 2017 году Illumina объединила технологию SBS с технологией CMOS, чтобы создать систему одноканального секвенирования.
- Использование датчика CMOS, встроенного в расходный материал, является простым и быстрым методом обнаружения.
- Система iSeqTM 100 использует одноканальную систему.
II. Секвенирование лигированием
Платформы для секвенирования легированием
|
Создатель
(производитель)
|
Амплификация
(увеличение числа копий участков ДНК)
|
Детекция
(система обнаружения)
|
|
SOLiD (ThermoFisher)
|
Клональная
|
Оптическая
|
|
Dover (Polonator)
|
Клональная
|
Оптическая
|
|
Complete Genomics
|
Клональная
|
Оптическая
|
Платформа SOLiD (Supported Oligonucleotide Ligation and Detection System 2.0), разработанная Applied Biosystems — технология секвенирования коротких чтений, основанная на лигировании (секвенирования ДНК с использованием ДНК-лигазы для создания новой комплементарной нити ДНК). По технологии SOLiD при секвенировании фрагменты ДНК лигируются на олигонуклеотидные адаптеры, прикрепленные к шарикам, далее они амплифицируются с помощью эмульсионной ПЦР.
III. Секвенирование с помощью нанопор
Платформы для нанопорного секвенирования
|
Создатель
(производитель)
|
Амплификация
(увеличение числа копий участков ДНК)
|
Детекция
(система обнаружения)
|
|
Oxford Nanopore
|
Одномолекулярная
|
Нанопоры
|
|
Roche Genia
|
Одномолекулярная
|
Нанопоры
|
|
Quantum Biosystems
|
Одномолекулярная
|
Нанопоры
|
Нанопоры - это поры в нанодиапазоне (~1-100 нм). Их можно разделить на три категории:
- Биологические - также называемые трансмембранными белковыми каналами, обычно вставляемыми в субстрат (мембрану). Четко определенные и высоко воспроизводимые размеры и структура нанопор.
- Твердотельные - синтетические нанопоры. Они имеют множество преимуществ перед своими биологическими аналогами, такие как химическая, термическая и механическая стабильность, возможность регулировки размера и интеграция.
- Гибрид - смесь обоих нанопор, использующая преимущества биологических и твердотельных нанопор.
Принцип детектирования основан на контроле ионного тока, проходящего через нанопору при подаче напряжения на мембрану.
Подробнее о Nanopore Sequencing
Нанопоровое секвенирование

Нанопоровое секвенирование - метод основан на измерении тока ионов через единичную нанопору в непроводящей мембране. При прохождении через эту пору нуклеотидов ток падает. Время, на которое изменяется ток ионов, и величина этого падения зависят от того, какой нуклеотид в данный момент находится внутри поры.
Нанопоровые системы представляют собой реакционную камеру, внутри которой находится раствор электролита. Камера разделена на две части липидной мембраной или иной тонкой непроводящей поверхностью, в которую внедрена единичная нанопора. К частям камеры прикладывают напряжение, из-за чего возникает ток ионов через пору. Когда исследуемые молекулы проходят через пору по направлению поля, они уменьшают сечение, доступное для ионов, и сила тока падает. Анализируя изменение силы тока, можно определить свойства молекулы, проходящей через пору.
В случае нуклеиновых кислот, диаметр используемых нанопор составляет несколько нанометров, из-за чего ДНК и РНК способны проходить сквозь пору только в одноцепочечной форме, но не в двухцепочечной. При прохождении молекулы нуклеиновой кислоты через пору отдельные нуклеотиды задерживаются в определенных сайтах внутри поры, в результате чего происходит измеримое падение силы тока.
Молекула ДНК или РНК, несущая на себе существенный отрицательный заряд, под воздействием электрического поля «протискивается» через белковую пору, помещенную в бислойную мембрану, что вызывает уменьшение силы электрического тока из-за уменьшения сечения отверстия. Считывая изменение силы тока, нанопоровый секвенатор определяет нуклеотид, проходящий через пору в конкретный отрезок времени: оказывается, что каждый «кирпичик» в нуклеиновой кислоте имеет свое собственное «сечение» (и, соответственно, это влияет на силу тока).
Технология секвенирования Nanopore позволяет делать прямое прочтение цепей ДНК или РНК в режиме онлайн, длина рида ограничена только длиной фрагмента, а портативность оборудования и быстрая подготовка библиотек дает возможность секвенировать даже в полевых условиях с минимальными требованиями к генетической лаборатории.
Нанопоровые секвенаторы ДНК 3-го поколения, не заменяют и не отменяют применение капиллярных секвенаторов по Сэнгеру или платформ NGS второго поколения. Наоборот, сочетание трех поколений генетического анализа открывает новые возможности получения ранее неизвестных данных
Итак, нанопоровое секвенирование предлагает:
- Прямое секвенирование нативной ДНК/РНК или образцов, которые были амплифицированы с помощью ПЦР / других методов
- Реальный результат в режиме реального времени
- Отсутствие требуемых капитальных затрат
- Считывание любой длины ДНК/РНК-от короткой до сверхдлинной
- Объем оборудования сводится до портативного или настольного компьютера
- Простая + быстрая + автоматизированная подготовка библиотек
- Высокиерезультаты для больших геномов
Биологические нанопоры
Рисунок 45. Биологические трансмембранные белковые каналы для нанопорного секвенирования
Примечание: Стоит отметить, что культуразависимые методы остаются незаменимыми для детальной идентификации бактерий. Тем не менее, успешное типирование бактерий вплоть до уровня видов/штаммов остается не полностью решенным. Однако нанопоровый молекулярный метод ON-rep-seq от Oxford Nanopore Technologies, при котором секвенируются бактериальные (или дрожжевые) селективные внутригенные фрагменты, полученные с помощью Rep-PCR, уже в 2019 г. позволил провести массовое типирование бактериальных видов до уровня штамма - см. подробне: Dennis S. Nielsen. DNA enrichment and tagmentation method for species-level identification and strain-level differentiation using ON-rep-seq. Communications Biology, 2019; 2 (1)
Об одном из прикладных значений секвенирования см. по кнопке-ссылке:
Материал в помощь:
 |
||
|
(Учебник содержит базовые знания по теме секвенирования нуклеиновых кислот)
|
(Темы междисциплинарной области знаний XXI века)
|
(Методы секвенирования 2-го и 3-го поколений)
|
Технологии секвенирования (краткий курс видеолекций)
Если видео не отображается, то оно доступно по ссылке→
Содержание видео:
• Введение в биоинформатику (0:00)
• Сиквенсные проекты (4:55)
• Алгоритмы сборки (8:43)
• Знакомство с методами выделения геномной ДНК (15:27)
• Определение количества и качества ДНК (29:16)
• Подготовка ДНК-библиотек для секвенатора Illumina MiSeq (36:53)
• Запуск секвенатора Illumina MiSeq (Illumina MiSeq sequencer running) (50:14)
• Основные понятия. Секвенирование по Сэнгеру (53:39)
• Обзор технологий секвенирования нового поколения (NGS) (1:02:32)
• Платформы для секвенирования: 454, Ion Torrent, SOLiD, Illumina (1:10:11)
• Synthetic Long Reads, PacBio, Oxford Nanopore (1:24:52)
• Сборка ДНК de novo - часть 1 (1:34:28)
• Сборка ДНК de novo - часть 2 (1:48:00)
Если Вам не хватает базовых знаний по молекулярной биологии, в т.ч. по основной терминологии и определениям, то все это можно найти в подборке "литература в помощь" в дополнительном материале раздела о ДНК прокариот и эукариот ↓
К разделу: ДНК прокариот и эукариот























